Linear Regression#
Universidad Central#
Maestría en analítica de datos#
Métodos estadísticos para analítica de datos.#
Docente: Luis Andrés Campos Maldonado.#
import folium
import warnings
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as mcolors
import seaborn as sns
import plotly.express as px
from sklearn.preprocessing import OneHotEncoder
from sklearn.model_selection import train_test_split # Train, Test sets
from sklearn.metrics import mean_absolute_error, r2_score # Para obtener las métricas de evaluación.
from sklearn.metrics import mean_absolute_percentage_error # Para obtener las métricas de evaluación.
from sklearn.linear_model import LinearRegression
# Parte estadística.
import statsmodels.formula.api as smf # Modelos lineal.
from scipy.stats import shapiro, probplot, f, f_oneway # libreria estadistica de Scipy
from statsmodels.stats.anova import anova_lm # Anova del modelo lineal
plt.style.use("ggplot")
plt.rcParams["figure.figsize"] = (15,6)
warnings.filterwarnings("ignore")
url_base = "https://raw.githubusercontent.com/lacamposm/Metodos-Estadisticos/main/data/"
Regresión lineal simple.#
La regresión lineal simple es una técnica estadística utilizada para modelar la relación entre dos variables: una dependiente y una independiente. La idea principal es encontrar una línea recta (llamada línea de regresión) que mejor se ajuste a los datos observados, de manera que podamos predecir los valores de la variable dependiente con base en los valores de la variable independiente.
El objetivo de la regresión lineal es minimizar la suma de los errores cuadráticos entre los valores observados \(y_i\) y los valores predichos \(\hat{y}_i\). La función de error (o función de costo) se puede expresar como:
Donde:
\(\text{RSS}\) es la suma de los residuos al cuadrado (residual sum of squares)
\(y_i\) es el valor observado de la variable dependiente.
\(\hat{y}_i = \hat{\beta}_0 + \hat{\beta}_1x_i\) es el valor predicho por la línea de regresión.
\(x_i\) es el valor de la variable independiente.
\(n\) es el número total de observaciones.
En regresión lineal simple, podemos encontrar los valores óptimos de los coeficientes (\beta_0) y (\beta_1) que minimizan la suma de los cuadrados de los residuos (RSS) mediante las siguientes fórmulas analíticas:
Consideremos la siguiente situación:
Se ha recolectado información sobre 122 individuos, cada uno de ellos respondió por:
Exposure: Numéro de años que el trabajador estuvo expuesto al polvo de algodón.PEFR: Proporciona una medida de la capacidad pulmonar del trabajador (tasa de flujo respiratorio).
¿Cómo se relaciona el PEFR con Exposure?
data = "LM_simple_example_1.csv"
df = pd.read_csv(url_base + data)
df.head()
| PEFR | Exposure | |
|---|---|---|
| 0 | 390 | 0 |
| 1 | 410 | 0 |
| 2 | 430 | 0 |
| 3 | 460 | 0 |
| 4 | 420 | 1 |
df.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 122 entries, 0 to 121
Data columns (total 2 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 PEFR 122 non-null int64
1 Exposure 122 non-null int64
dtypes: int64(2)
memory usage: 2.0 KB
Primero veamos la relación vía un scatterplot:
(
px.scatter(
df, x="Exposure", y="PEFR", template="plotly_white",
title=f"Scatterplot: Exposure vs. PEFR.<br>Coeficiente de correlacion de Pearson: {df.corr().iloc[0,1]:.3f}"
)
).show()
Recordemos que la regresión lineal simple estima cuánto cambiará PERF cuando Exposure cambie en un
cierta cantidad. Es decir, deseamos explicar si las diferencias en los niveles de PERF se asocian con las diferencias en los años de exposición (Exposure). Nuestro modelo será en este caso:
\(\beta_0\) es una constante conocida como
el intercepto, \(\beta_1\) será la pendiente, ambos de denominan parámetros de la regresión. \(\epsilon\) es un error o ruido que NO se logra explicar con el modelo. La regresión lineal simple trata de encontrar la “mejor” recta para predecir la variable Target PEFR como una función de la feature Exposure
Hacemos uso de sklearn
# Ajuste de regresión con sklearn
model_lm = LinearRegression()
y = df["PEFR"] # Target.
X = df.drop(columns=["PEFR"]) # Feature.
model_lm.fit(X, y)
LinearRegression()In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
LinearRegression()
print(f"Intercepto: {model_lm.intercept_:.3f}")
print(f"Coeficiente Exposure: {model_lm.coef_[0]:.3f}")
Intercepto: 424.583
Coeficiente Exposure: -4.185
En caso que seleccionemos este ajuste muestro modelo será:
La interpretación de los coeficientes es la siguiente:
\(\hat{\beta_0}=424.583\): Es el intercepto del modelo, note que para un valor de \(Exposure=0\), se obtiene que \(PEFR=424.583\), así, se espera que un trabajador sin exposición tenga ese valor medio del indicador de respiración.
\(\hat{\beta_1}=-4.185\): Por cada aumento unitario en la variable \(Exposure\) se espera que \(PEFR\) disminuya en promedio el valor en \(\hat{\beta_1}\)
Veamos la recta de ajuste:
# Regresión simple, recta de ajuste por OLS.
px.scatter(
df, x="Exposure", y="PEFR", template="plotly_white",
title="Recta de ajuste por OLS", trendline="ols",
trendline_color_override="red"
).show()
Valores ajustados y residuales.#
Los conceptos importantes en el análisis de regresión son los valores ajustados (las predicciones con el modelo) y los residuales (errores de predicción). En general, para \(Y\) variable target y \(X\) variable explicativa o (feature para predicir), los datos no caen exactamente en la recta de ajuste, por lo que la ecuación de regresión debe incluir un término de error explícito \(\epsilon_i\)
Los valores ajustados, denotados por \(\hat{Y_i}\) vienes dados por la expresión:
Calculamos los residuos estimados (\(\hat{\epsilon_i}\)) mediante:
fitted = model_lm.predict(X) # Valores ajustados Yi^
resid = y - fitted # Residuales estimados.
# Residuales
pd.DataFrame({"valor_observado":y, "valor_ajustado":fitted,"residual":resid})
| valor_observado | valor_ajustado | residual | |
|---|---|---|---|
| 0 | 390 | 424.582807 | -34.582807 |
| 1 | 410 | 424.582807 | -14.582807 |
| 2 | 430 | 424.582807 | 5.417193 |
| 3 | 460 | 424.582807 | 35.417193 |
| 4 | 420 | 420.398230 | -0.398230 |
| ... | ... | ... | ... |
| 117 | 450 | 332.522124 | 117.477876 |
| 118 | 490 | 332.522124 | 157.477876 |
| 119 | 500 | 332.522124 | 167.477876 |
| 120 | 370 | 328.337547 | 41.662453 |
| 121 | 390 | 328.337547 | 61.662453 |
122 rows × 3 columns
Anova#
Dado que queremos explicar la variabilidad de la variable Target con respecto a su media podemos descomponerla de la siguiente manera:
En la expresión expresión anterior se identifican:
Suma de cuadrados totales: $\(SCT = \sum_{i=1}^{n}(y_i-\bar{y})^{2}\)$ Esta suma determina la variabilidad respecto a su media.
Suma de cuadrados del error: $\(SCE=\sum_{i=n}^{n}(y_i-\hat{y_i})^{2}\)$ Esta suma toma en cuenta la variación con la recta estimada. La variación residual que queda sin explicar por la recta de regresión.
Suma de cuadrados de la regresión: $\(SCR=\sum_{i=1}^{n}(\hat{y_i}-\bar{y})^{2}\)$ La cantidad de variabilidad en las observaciones explicada por la recta regresión. Esta suma toma en cuenta la variación de las observaciones y es atribuible al efecto lineal del modelo.
# With statsmodels. from statsmodels.stats.anova import anova_lm ---> Anova del modelo lineal
model_lm_sm = smf.ols(formula="PEFR ~ Exposure", data=df).fit() ## Ajuste con statsmodels.
display(model_lm_sm.summary())
print("\nAnova:")
anova_lm(model_lm_sm, typ=1)
| Dep. Variable: | PEFR | R-squared: | 0.077 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.069 |
| Method: | Least Squares | F-statistic: | 9.974 |
| Date: | Thu, 05 Dec 2024 | Prob (F-statistic): | 0.00201 |
| Time: | 14:56:32 | Log-Likelihood: | -735.68 |
| No. Observations: | 122 | AIC: | 1475. |
| Df Residuals: | 120 | BIC: | 1481. |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 424.5828 | 20.796 | 20.417 | 0.000 | 383.408 | 465.757 |
| Exposure | -4.1846 | 1.325 | -3.158 | 0.002 | -6.808 | -1.561 |
| Omnibus: | 0.767 | Durbin-Watson: | 1.111 |
|---|---|---|---|
| Prob(Omnibus): | 0.681 | Jarque-Bera (JB): | 0.891 |
| Skew: | -0.162 | Prob(JB): | 0.641 |
| Kurtosis: | 2.734 | Cond. No. | 35.7 |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Anova:
| df | sum_sq | mean_sq | F | PR(>F) | |
|---|---|---|---|---|---|
| Exposure | 1.0 | 1.026333e+05 | 102633.255269 | 9.974366 | 0.002008 |
| Residual | 120.0 | 1.234764e+06 | 10289.702381 | NaN | NaN |
La tabla anterior es una típica ANOVA.
El estadístico \(F_{1}^{n-2}\) se utiliza para contrastar el sistema de hipótesis:
Es decir, este mide la pertinencia o no del modelo (Significancia global del modelo).
Para el caso específico de nuestro problema el \(p\)-valor de la prueba es \(0.002008\), es decir, podemos explicar las diferencias en la variable PEFR con las diferencias existentes en la variable Exposure.
Coeficiente de determinación.#
El coeficiente de determinación se encuentra con la fórmula:
Al ser \(SCT\) una medida de la variabilidad de \(Y\) sin considerar el efecto de la variable regresora y \(SCE\) es una medida de la variabilidad de \(Y\) que queda después de haber tenido en consideración a \(X\), el coeficiente de determinación, \(R^{2}\) se llama con frecuencia la proporción de la variación explicada por el regresor \(X\). Este valor va de 0 y hasta 1, valores cercanos a 1 implican que la mayor parte de la variabilidad de \(Y\) está explicada por el modelo de regresión.
SCE = 1.234764e+06 # Suma de cuadrados del error.
SCR = 1.026333e+05 # Suma de cuadrados de la regresión.
SCT = (1.234764e+06+1.026333e+05)
R_2 = 1- SCE/SCT
print("El valor de R^2 es: {:.4f}".format(R_2))
print("Por lo tanto el {:.2f}% de la variabilidad de PEFR queda explicada por el modelo.".format(R_2*100))
#
print()
print(f"R^2 Con sklearn: {model_lm.score(X,y):.4f}")
# Con statsmodels
print()
print("Suma de cuadrados del error:", model_lm_sm.ssr)
print("Suma de cuadrados de la regresión:", model_lm_sm.mse_model)
print("Suma de cuadrados totales:", model_lm_sm.centered_tss)
print("R^2 con statsmodels:", model_lm_sm.rsquared)
El valor de R^2 es: 0.0767
Por lo tanto el 7.67% de la variabilidad de PEFR queda explicada por el modelo.
R^2 Con sklearn: 0.0767
Suma de cuadrados del error: 1234764.285714286
Suma de cuadrados de la regresión: 102633.25526932045
Suma de cuadrados totales: 1337397.5409836064
R^2 con statsmodels: 0.07674102286283369
Análisis de residuales#
Debemos verificar que los residuales no estén correlacionados.
Homocedasticidad.
Que los residuales distribuyan aproximadamente normal
En primera medida debemos estimar el valor de \(\sigma^{2}\), este valor está dado por la expresión:
Que no es otra cosa que el cuadrado medio del error.
# Estimación de sigma^2.
sigma_2 = 10289.702381
print("La estimación de la variabilidad del modelo es:", sigma_2)
# Estimación de sigma^2 con statsmodels.
print("La estimación de sigma^2 con statsmodels es:", model_lm_sm.scale)
La estimación de la variabilidad del modelo es: 10289.702381
La estimación de sigma^2 con statsmodels es: 10289.702380952383
Normalidad.#
# NORMALIDAD: Test de Shapiro-Wills
# La prueba de Shapiro-Wilk prueba la hipótesis nula de que los datos se extrajeron
# de una distribución normal.
p_value1 = shapiro(df["PEFR"])[1] ### Normalidad variable respuesta
print("El p-valor del Test de Shapiro-Wills para la variable respuesta es: {:.3f}".format(p_value1))
p_value2 = shapiro(resid)[1]
print("El p-valor del Test de Shapiro-Wills para normalidad de los residuales:{:.3f}".format(p_value2))
El p-valor del Test de Shapiro-Wills para la variable respuesta es: 0.541
El p-valor del Test de Shapiro-Wills para normalidad de los residuales:0.586
Luego no podemos rechazar la hipótesis nula, por lo tanto no tenemos evidencia estadística para decir que los residuales no distribuyen aproximadamente normal.
# QQ plot para los residuales observados. from scipy.stats import probplot
plt.figure(figsize = (15,4))
probplot(x=resid, dist="norm", plot=plt) ## QQ-plot.
plt.xlabel("Cuantiles teóricos")
plt.ylabel("Valores ordenados")
plt.title("QQ Plot para los residuales.")
plt.show()
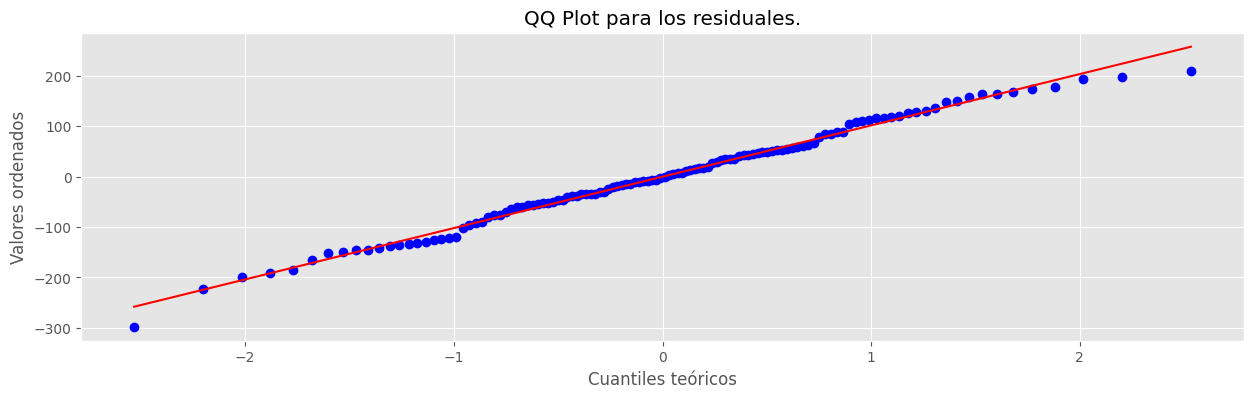
Como conclusión notemos que se cumplen los supuestos del modelo, pero el poder explicativo del modelo solo llega al \(7.67\%\), lo cual no es un buen indicador.
Regresión lineal múltiple.#
En una regresión lineal múltiple, el objetivo es modelar la relación entre una variable dependiente \(Y\) (variable objetivo o Target) y múltiples variables explicativas \(X_1, X_2, \ldots, X_p\), de tal manera que se pueda predecir el comportamiento de \(Y\) en función de estas variables explicativas. Entonces se quiere construir una función \(f(X)\) donde \(X=(X_1, \ldots, X_p)\) es un conjunto de variables explicativas (Features) que se utilizaran para pronosticar y explicar las diferencias presentes en la variable target \(Y\).
En un modelo de regresión lineal, se usan las funciones del tipo: $\(Y=\beta_0 +\beta_1X_1+\beta_2X_2+...+\beta_pX_p +\epsilon \)$
donde \(\epsilon\) se conoce como el error o ruido del modelo.
Sobre este error se realizan varios supuestos para que el modelo tenga validez estadística.
Normalidad o gaussianidad: Campana de Gauss
Homocedasticidad: La variabilidad de mi modelo no depende de las X
Independencia.
La funcion a optimizar en este se toma la forma:
Donde:
\(\text{RSS}\) es la suma de los residuos al cuadrado (Residual Sum of Squares).
\(y_i\) es el valor observado de la variable dependiente \(Y\).
\(X_{i1}, X_{i2}, \dots, X_{ip}\) son los valores de las variables explicativas para la \(i\)-ésima observación.
La estimación de los coeficientes \(\beta_0, \beta_1, \dots, \beta_p\) que minimizan la función de costo se puede obtener mediante la fórmula de mínimos cuadrados ordinarios (OLS) en su forma matricial:
Ejemplo 1.#
Con el objetivo de evaluar la calidad de una película utilizada en máquinas fotográficas, el tiempo de duración de la película, en horas, se relaciona con la densidad máxima de la película bajo tres condiciones experimentales: \(72^{o}C\), \(82^{o}C\) y \(92^{o}C\). El objetivo es explicar las diferencias en el tiempo de duración de la película usando la densidad máxima y la temperatura.
df2 = pd.read_excel(url_base + "chapman.xlsx")
df2.head()
df2.describe().T
| count | mean | std | min | 25% | 50% | 75% | max | |
|---|---|---|---|---|---|---|---|---|
| tiempo | 21.0 | 192.000000 | 133.194594 | 24.00 | 96.00 | 144.00 | 288.00 | 504.00 |
| densidad | 21.0 | 2.264762 | 0.890020 | 0.65 | 1.49 | 2.27 | 2.91 | 3.55 |
| temperatura | 21.0 | 82.000000 | 8.366600 | 72.00 | 72.00 | 82.00 | 92.00 | 92.00 |
Haga un gráfico de los datos que incluya las tres variables consideradas. Comente.
df2["temperatura"] = df2["temperatura"].astype("object")
px.scatter(
df2, x="densidad", y="tiempo", template="plotly_white",
title="Densidad vs. Tiempo", trendline="ols", trendline_color_override="red"
).show()
Respuesta
Los plots anterior muestra que el tiempo de duración de una película disminuye a medida que la densidad máxima de la película aumenta, es decir, al parecer existe una relación lineal negativa entre el Tiempo de duración de la película y la densidad máxima de la película. Además es diferente según las tres condiciones experiementales, quizá se presenta interacción entre densidad y temperatura.
px.scatter(
df2, x="densidad", y="tiempo", color="temperatura",
template="plotly_white", title="Densidad vs. Tiempo", trendline="ols"
).show()
Ejemplo 2.#
Ajuste a los datos un modelo de regresión lineal múltiple donde la respuesta es el tiempo de duración de la película y las variables explicativas son la densidad máxima, la condición experimental y la interacción entre estas.
# Media de cada grupo de "temperatura" de la variable Target "tiempo".
df2.groupby("temperatura")["tiempo"].mean()
temperatura
72 288.0
82 192.0
92 96.0
Name: tiempo, dtype: float64
# Modelo con solo la feature categórica.
cate = smf.ols(formula="tiempo ~ temperatura", data=df2).fit()
print(cate.summary())
OLS Regression Results
==============================================================================
Dep. Variable: tiempo R-squared: 0.364
Model: OLS Adj. R-squared: 0.293
Method: Least Squares F-statistic: 5.143
Date: Thu, 05 Dec 2024 Prob (F-statistic): 0.0171
Time: 14:56:32 Log-Likelihood: -127.27
No. Observations: 21 AIC: 260.5
Df Residuals: 18 BIC: 263.7
Df Model: 2
Covariance Type: nonrobust
=====================================================================================
coef std err t P>|t| [0.025 0.975]
-------------------------------------------------------------------------------------
Intercept 288.0000 42.332 6.803 0.000 199.064 376.936
temperatura[T.82] -96.0000 59.867 -1.604 0.126 -221.775 29.775
temperatura[T.92] -192.0000 59.867 -3.207 0.005 -317.775 -66.225
==============================================================================
Omnibus: 0.007 Durbin-Watson: 0.995
Prob(Omnibus): 0.997 Jarque-Bera (JB): 0.123
Skew: 0.000 Prob(JB): 0.940
Kurtosis: 2.625 Cond. No. 3.73
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Para este caso se observa que la diferencia de las medias es estadísticamente significativa, es decir, hay diferencias en las medias entre las categorías.
# Modelo SIN interacción.
model_ex2_with_inter = smf.ols(formula="tiempo ~ densidad + temperatura", data=df2).fit()
print(model_ex2_with_inter.summary())
OLS Regression Results
==============================================================================
Dep. Variable: tiempo R-squared: 0.796
Model: OLS Adj. R-squared: 0.760
Method: Least Squares F-statistic: 22.15
Date: Thu, 05 Dec 2024 Prob (F-statistic): 4.15e-06
Time: 14:56:32 Log-Likelihood: -115.31
No. Observations: 21 AIC: 238.6
Df Residuals: 17 BIC: 242.8
Df Model: 3
Covariance Type: nonrobust
=====================================================================================
coef std err t P>|t| [0.025 0.975]
-------------------------------------------------------------------------------------
Intercept 568.0902 52.724 10.775 0.000 456.853 679.327
temperatura[T.82] -133.5269 35.407 -3.771 0.002 -208.229 -58.825
temperatura[T.92] -275.0737 37.493 -7.337 0.000 -354.178 -195.969
densidad -105.9228 17.627 -6.009 0.000 -143.111 -68.734
==============================================================================
Omnibus: 0.449 Durbin-Watson: 0.617
Prob(Omnibus): 0.799 Jarque-Bera (JB): 0.338
Skew: 0.278 Prob(JB): 0.845
Kurtosis: 2.723 Cond. No. 11.9
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Con el modelo anterior se logra explicar un \(79.6\%\) de las diferennias de la variable Target (tiempo). Note además que con este modelo obtemos una expresión de la forma:
En cada obtenemos una recta de regresión:
Caso 1: Corresponde a la temperatura de 72 grados: \(tiempo=568.09-105.9228*densidad\):
Caso 2: Corresponde a la temperatura de 82 grados: \(tiempo=434.57 -105.922* densidad\)
Caso 3: Corresponde a la temperatura de 92 grados: \(tiempo=293.016 -105.922* densidad\)
Estas rectas tienen la particularidad de que son rectas paralelas, es decir, no estamos teniendo en cuenta la interacción entre las dos 2 features.
Modelado de efectos de interacción.
# Modelo CON interacción.
model_ex2 = smf.ols(formula="tiempo ~ densidad*temperatura", data=df2).fit()
print(model_ex2.summary())
OLS Regression Results
==============================================================================
Dep. Variable: tiempo R-squared: 0.989
Model: OLS Adj. R-squared: 0.985
Method: Least Squares F-statistic: 266.3
Date: Thu, 05 Dec 2024 Prob (F-statistic): 4.30e-14
Time: 14:56:32 Log-Likelihood: -84.791
No. Observations: 21 AIC: 181.6
Df Residuals: 15 BIC: 187.8
Df Model: 5
Covariance Type: nonrobust
==============================================================================================
coef std err t P>|t| [0.025 0.975]
----------------------------------------------------------------------------------------------
Intercept 936.5894 28.420 32.956 0.000 876.014 997.165
temperatura[T.82] -482.0048 33.706 -14.300 0.000 -553.848 -410.162
temperatura[T.92] -750.2118 31.380 -23.907 0.000 -817.096 -683.327
densidad -245.2796 10.494 -23.373 0.000 -267.647 -222.912
densidad:temperatura[T.82] 130.6139 12.868 10.150 0.000 103.187 158.041
densidad:temperatura[T.92] 196.6895 12.264 16.037 0.000 170.549 222.830
==============================================================================
Omnibus: 0.398 Durbin-Watson: 1.731
Prob(Omnibus): 0.820 Jarque-Bera (JB): 0.537
Skew: -0.175 Prob(JB): 0.765
Kurtosis: 2.300 Cond. No. 43.7
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Ejercicio 5.#
Interprete las estimaciones de los parámetros excepto el intercepto.
px.scatter(
df2, x="densidad", y="tiempo", color="temperatura",
template="plotly_white", title="Densidad vs. Tiempo", trendline="ols"
).show()
Ejercicio 6.#
Estime la duración esperada de una película con una densidad máxima de 2, y una temperatura de \(82^{o}C\). Use el método predict() del modelo ajustado.
model_ex2.predict({"densidad": 2, "temperatura": 82})
0 225.253073
dtype: float64
Comentario.
El tiempo esperado de duración de una película bajo la condición experimental de \(82^{o}C\) y con densidad máxima igual a 2 es de \(225.2531\).
Ejemplo 3#
df = pd.read_csv(url_base + "kc_house_data.csv")
df.head()
| id | date | price | bedrooms | bathrooms | sqft_living | sqft_lot | floors | waterfront | view | ... | grade | sqft_above | sqft_basement | yr_built | yr_renovated | zipcode | lat | long | sqft_living15 | sqft_lot15 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 7129300520 | 20141013T000000 | 221900.0 | 3 | 1.00 | 1180 | 5650 | 1.0 | 0 | 0 | ... | 7 | 1180 | 0 | 1955 | 0 | 98178 | 47.5112 | -122.257 | 1340 | 5650 |
| 1 | 6414100192 | 20141209T000000 | 538000.0 | 3 | 2.25 | 2570 | 7242 | 2.0 | 0 | 0 | ... | 7 | 2170 | 400 | 1951 | 1991 | 98125 | 47.7210 | -122.319 | 1690 | 7639 |
| 2 | 5631500400 | 20150225T000000 | 180000.0 | 2 | 1.00 | 770 | 10000 | 1.0 | 0 | 0 | ... | 6 | 770 | 0 | 1933 | 0 | 98028 | 47.7379 | -122.233 | 2720 | 8062 |
| 3 | 2487200875 | 20141209T000000 | 604000.0 | 4 | 3.00 | 1960 | 5000 | 1.0 | 0 | 0 | ... | 7 | 1050 | 910 | 1965 | 0 | 98136 | 47.5208 | -122.393 | 1360 | 5000 |
| 4 | 1954400510 | 20150218T000000 | 510000.0 | 3 | 2.00 | 1680 | 8080 | 1.0 | 0 | 0 | ... | 8 | 1680 | 0 | 1987 | 0 | 98074 | 47.6168 | -122.045 | 1800 | 7503 |
5 rows × 21 columns
Exploración de datos#
Comencemos por revisar las columnas del conjunto de datos y lo que significan:
id: Identificación para la casa.
date: Fecha de venta de la casa.
price: Precio al que se vendió la casa.
bedrooms: Número de habitaciones.
bathrooms: Número de baños.
sqft_living: Pies cuadrados de la casa.
sqft_lot: Pies cuadrados del lote.
floors: Total de pisos (niveles) en casa.
waterfront: Si la casa tiene o no una vista de un paseo marítimo (whether or not the house has a view of a waterfront)
view: Un índice de 0 a 4 de qué tan buena es la vista desde la propiedad
condition: Condición de la casa (que tan buena está).
grade: Calificación general otorgada a la unidad de vivienda, basada en el sistema de calificación del condado de King.
sqft_above: Metros cuadrados de la casa aparte del sótano
sqft_basement: Metros cuadrados del sótano
yr_built: Año en que se construyó la casa
yr_renovated: Año en que fué renovada la casa.
zipcode: Código postal de la casa
lat: latitude coordinate of the house
long: longitude coordinate of the house
sqft_lot15 :lotSize area in 2015(implies– some renovations)
sqft_living15: Área de la sala de estar en 2015 (implica: algunas renovaciones) Esto podría o no haber afectado el área del tamaño del lote
# Vamos a formar nuestro Dataframe de trabajo
df_ml = df.drop(columns=["id", "sqft_living15", "sqft_lot15"])
df_ml.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 21613 entries, 0 to 21612
Data columns (total 18 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 date 21613 non-null object
1 price 21613 non-null float64
2 bedrooms 21613 non-null int64
3 bathrooms 21613 non-null float64
4 sqft_living 21613 non-null int64
5 sqft_lot 21613 non-null int64
6 floors 21613 non-null float64
7 waterfront 21613 non-null int64
8 view 21613 non-null int64
9 condition 21613 non-null int64
10 grade 21613 non-null int64
11 sqft_above 21613 non-null int64
12 sqft_basement 21613 non-null int64
13 yr_built 21613 non-null int64
14 yr_renovated 21613 non-null int64
15 zipcode 21613 non-null int64
16 lat 21613 non-null float64
17 long 21613 non-null float64
dtypes: float64(5), int64(12), object(1)
memory usage: 3.0+ MB
# "waterfront", "zipcode","view" no estan en el tipo de dato adecuado,
df_ml[["waterfront", "zipcode", "view"]] = df_ml[["waterfront", "zipcode", "view"]].astype("category")
df_ml.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 21613 entries, 0 to 21612
Data columns (total 18 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 date 21613 non-null object
1 price 21613 non-null float64
2 bedrooms 21613 non-null int64
3 bathrooms 21613 non-null float64
4 sqft_living 21613 non-null int64
5 sqft_lot 21613 non-null int64
6 floors 21613 non-null float64
7 waterfront 21613 non-null category
8 view 21613 non-null category
9 condition 21613 non-null int64
10 grade 21613 non-null int64
11 sqft_above 21613 non-null int64
12 sqft_basement 21613 non-null int64
13 yr_built 21613 non-null int64
14 yr_renovated 21613 non-null int64
15 zipcode 21613 non-null category
16 lat 21613 non-null float64
17 long 21613 non-null float64
dtypes: category(3), float64(5), int64(9), object(1)
memory usage: 2.5+ MB
Variable Target#
px.histogram(df_ml["price"], title="Distribucion variable objetivo", template="plotly_white").show()
# Shapiro Test.
shapiro(df_ml["price"])
# No hay normalidad...
ShapiroResult(statistic=0.7120112962695346, pvalue=8.944594546946854e-104)
# QQ plot de la variable Target ----> "price"
plt.figure(figsize = (15,4))
probplot(x=df_ml["price"], dist="norm", plot=plt)
plt.xlabel("Cuantiles teóricos")
plt.ylabel("Valores ordenados")
plt.title("QQ Plot para los precios.")
plt.show()
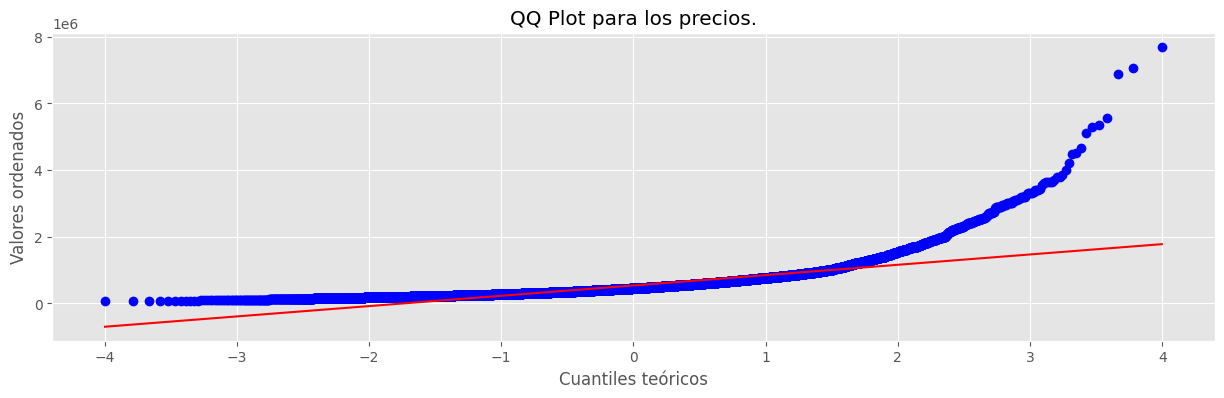
Notemos que la distribución se aleja bastante de la distribución normal. El algunos casos podemos buscar normalizar la variable Target. Recuerde que puede usar la transformación de Box-Cox
# Note que podemos hacer uso de la siguiente transformación: (Coincide con Box-Cox!!!)
px.histogram(x=np.log(df_ml["price"]), title="Logaritmo natural de la variable price, log(price)", template="plotly_white")
# Transformamos la varible target, log(price)
df_ml["log_price"] = np.log(df_ml["price"])
# Veamos las correlaciones.
sns.heatmap(df_ml.drop(columns=["price"]).select_dtypes(np.number).corr(), annot=True)
plt.show()
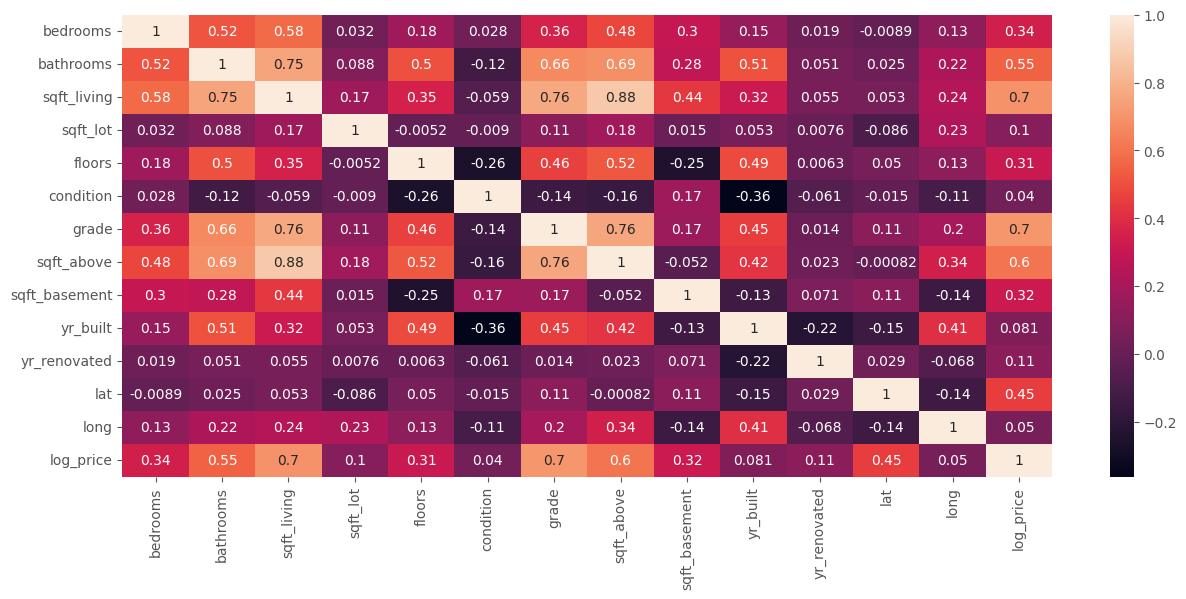
# Tomemos un umbral pues tenemos bastantes variables
higt_corr = df_ml.drop(columns=["price"]).select_dtypes(np.number).corr().loc["log_price"]
higt_corr.sort_values(ascending=False)
log_price 1.000000
grade 0.703634
sqft_living 0.695341
sqft_above 0.601801
bathrooms 0.550802
lat 0.449174
bedrooms 0.343561
sqft_basement 0.316970
floors 0.310558
yr_renovated 0.114498
sqft_lot 0.099622
yr_built 0.080655
long 0.049942
condition 0.039558
Name: log_price, dtype: float64
# Exploración con una de las más altamente relacionada
px.scatter(
df_ml, x="sqft_living", y="log_price", title="Sqft_living vs. Precio",
trendline="ols", trendline_color_override="red", template="plotly_white"
).update_traces(marker=dict(size=3)).show()
df_temp = df_ml[["log_price", "sqft_living"]]
df_temp["log_sqft_living"] = np.log(df_temp["sqft_living"])
px.scatter(
df_temp, x="log_sqft_living", y="log_price", title="Sqft_living vs. Precio (log-log)",
trendline="ols", trendline_color_override="red", template="plotly_white"
).update_traces(marker=dict(size=3))
Modelo Log-Log.#
El primer modelo de inspección es:
formula = "log_price~log_sqft_living"
log_log_model = smf.ols(formula, data=df_temp).fit()
print(log_log_model.summary())
OLS Regression Results
==============================================================================
Dep. Variable: log_price R-squared: 0.456
Model: OLS Adj. R-squared: 0.455
Method: Least Squares F-statistic: 1.808e+04
Date: Thu, 05 Dec 2024 Prob (F-statistic): 0.00
Time: 14:56:33 Log-Likelihood: -10240.
No. Observations: 21613 AIC: 2.048e+04
Df Residuals: 21611 BIC: 2.050e+04
Df Model: 1
Covariance Type: nonrobust
===================================================================================
coef std err t P>|t| [0.025 0.975]
-----------------------------------------------------------------------------------
Intercept 6.7299 0.047 143.001 0.000 6.638 6.822
log_sqft_living 0.8368 0.006 134.459 0.000 0.825 0.849
==============================================================================
Omnibus: 123.344 Durbin-Watson: 1.978
Prob(Omnibus): 0.000 Jarque-Bera (JB): 113.759
Skew: 0.142 Prob(JB): 1.98e-25
Kurtosis: 2.787 Cond. No. 137.
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
Tenemos que ser conscientes de cómo interpretamos los coeficientes aquí. En una regresión en la que tanto la variable independiente como la dependiente son transformaciones logarítmicas (una log-log regression), el coeficiente \(\beta_1\) debe interpretarse como el cambio porcentual en la variable dependiente asociada con el 1% de cambio en la variable independiente. Esta comparación de cambio de porcentaje frente a porcentaje se conoce como elasticidad. Así, en nuestro modelo:
Un aumento del 1% en la superficie habitable se asocia con un aumento del 0,8368% en el precio.
Modelo Log-Level#
Ahora construyamos un modelo lineal donde la transformación logarítmica solo se aplica a los precios de la vivienda, es decir, un modelo de la forma: $\( \log(price) = \beta_0 + \beta_1 sqft{\_}living + \varepsilon \)$
El plot y la tabla de salida están a continuación:
log_log_model = smf.ols("log_price ~ sqft_living", data=df_ml).fit()
print(log_log_model.summary())
OLS Regression Results
==============================================================================
Dep. Variable: log_price R-squared: 0.483
Model: OLS Adj. R-squared: 0.483
Method: Least Squares F-statistic: 2.023e+04
Date: Thu, 05 Dec 2024 Prob (F-statistic): 0.00
Time: 14:56:33 Log-Likelihood: -9670.2
No. Observations: 21613 AIC: 1.934e+04
Df Residuals: 21611 BIC: 1.936e+04
Df Model: 1
Covariance Type: nonrobust
===============================================================================
coef std err t P>|t| [0.025 0.975]
-------------------------------------------------------------------------------
Intercept 12.2185 0.006 1916.883 0.000 12.206 12.231
sqft_living 0.0004 2.8e-06 142.233 0.000 0.000 0.000
==============================================================================
Omnibus: 3.128 Durbin-Watson: 1.979
Prob(Omnibus): 0.209 Jarque-Bera (JB): 3.149
Skew: 0.027 Prob(JB): 0.207
Kurtosis: 2.974 Cond. No. 5.63e+03
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 5.63e+03. This might indicate that there are
strong multicollinearity or other numerical problems.
La interpretación del coeficiente de regresión vuelve a ser diferente. Esta es una log-level regression; es decir, uno en el que la variable dependiente se ha transformado logarítmicamente y la variable independiente no. Interpretamos el coeficiente como una semi-elasticidad, donde un aumento absoluto en sqft_living (porque no se le ha aplicado la función logarítmica) corresponde a un aumento porcentual en price. Específicamente, aquí podemos decir que:
Un aumento en el espacio habitable de 1 pie cuadrado conduce a un aumento del 0,04 % en el precio.
Podemos ver en estos gráficos que los puntos de datos del modelo log-log se agrupan más uniformemente alrededor de la línea de mejor ajuste en diferentes niveles de la variable independiente en comparación con el otro modelo, lo que sugiere que el modelo log-log es más lineal.
Corriendo regresiones con variables transformadas.#
Por supuesto, no tiene sentido limitarnos a modelar los precios de la vivienda usando solo una variable independiente. Agreguemos varias variables más, algunas transformadas y otras no.
¿En el contexto del problema que otras features considera importantes?. Podemos pensar en:
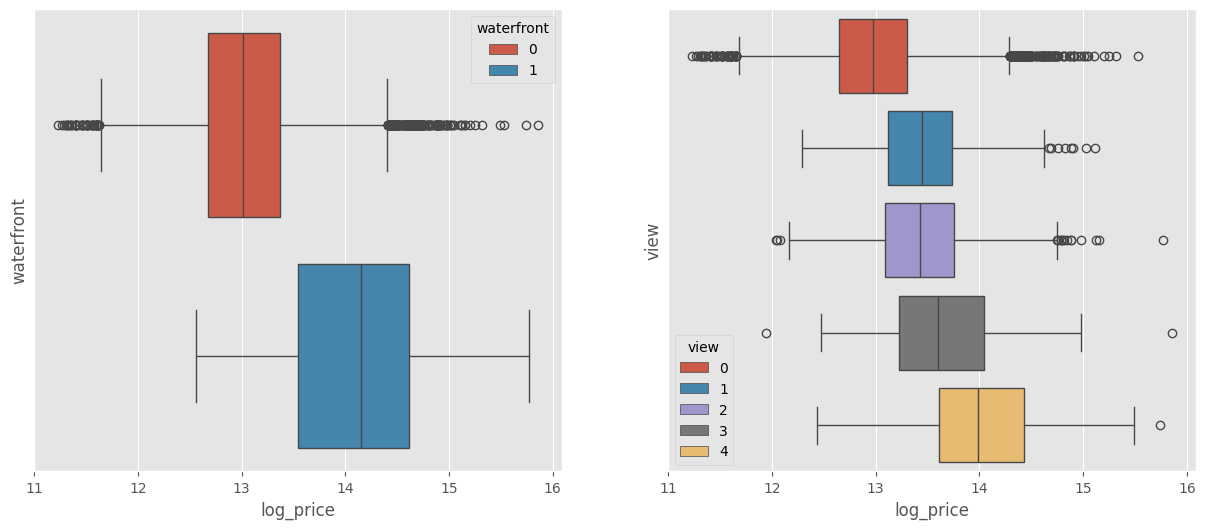
zipcode(Ubicación del predio ).waterfroont(Cercania al mar).view(Vista del predio)
# Sirven para discriminar??
fig, ax = plt.subplots(1,2)
sns.boxplot(data=df_ml, x="log_price", y="waterfront", ax=ax[0], hue="waterfront")
sns.boxplot(data=df_ml, x="log_price", y="view", ax=ax[1], hue="view")
ax[0].set_yticks([])
ax[0].set_yticklabels([])
ax[1].set_yticks([])
ax[1].set_yticklabels([])
plt.show()
sns.boxplot(data=df_ml, x="log_price", y="zipcode")
plt.show()
Vamos a incorporarlas al modelo.
Hasta este momento hemos tenido en cuenta las siguientes features para incluir en el modelo:
log_sqft_living(sqft_living).zipcode.viewwaterfrontgrade.sqft_above
Contextualizacion geografica.#
# Contextualizacion Geografica.
cmap = mcolors.LinearSegmentedColormap.from_list("custom_cmap", colors=["yellow", "red"], N=9)
map = folium.Map(
location=[df_ml["lat"].mean(), df_ml["long"].mean()],
zoom_start=9,
min_zoom=4
)
title_html = """
<h2 align="center" style="font-size:25px"><b>Color Map</b></h2>
"""
map.get_root().html.add_child(folium.Element(title_html))
color_scale = folium.LinearColormap(
colors=[mcolors.rgb2hex(cmap(i)) for i in range(9)],
vmin=df_ml["price"].quantile(0.01), vmax=df_ml["price"].quantile(0.99)
)
for index, row in df_ml.iterrows():
folium.CircleMarker(
location=[row["lat"], row["long"]],
radius=0.001,
color=color_scale(row["price"]),
fill=True,
fill_color=color_scale(row["price"]),
popup=f"price: ${int(row['price'])}"
).add_to(map)
color_scale.add_to(map)
map
# Veamos el comportamiento de "lat"
(
px.scatter(
df_ml, x="lat", y="log_price", trendline="ols",
trendline_color_override="red",
template="plotly_white"
)
.update_traces(marker=dict(size=2, color="blue"))
.show()
)
Note que existe cierta relación lineal entre la variable Target y lat, además, puede observar lo siguiente:
(
px.scatter(
df_ml, x="lat", y="log_price", trendline="ols",
template="plotly_white", color="waterfront"
)
.update_traces(marker=dict(size=2.5))
.show()
)
El plot anterior sugiere que podemos ingresar lat además del efecto de la interacción
# Consideremos variables que no estén incluidas
temp = pd.DataFrame(
{
"log_sqft_lot": np.log(df_ml["sqft_lot"]),
"log_price": df_ml["log_price"]
}
)
px.scatter(
temp, x="log_sqft_lot", y="log_price", trendline="ols",
trendline_color_override="red",
template="plotly_white"
).update_traces(marker=dict(size=1.8, color="blue")).show()
df_ml["renovated"] = df_ml["yr_renovated"] > 0
formula_ref = """
np.log(price) ~ np.log(sqft_living)*C(renovated) + np.log(sqft_lot)
+ bedrooms + floors + bathrooms + condition + C(view) + grade + yr_built
+ lat*C(waterfront) + I(lat**2) + long + C(zipcode)
"""
model_ref = smf.ols(formula=formula_ref, data=df_ml).fit()
print(model_ref.summary())
OLS Regression Results
==============================================================================
Dep. Variable: np.log(price) R-squared: 0.880
Model: OLS Adj. R-squared: 0.880
Method: Least Squares F-statistic: 1795.
Date: Thu, 05 Dec 2024 Prob (F-statistic): 0.00
Time: 14:56:46 Log-Likelihood: 6111.7
No. Observations: 21613 AIC: -1.205e+04
Df Residuals: 21524 BIC: -1.134e+04
Df Model: 88
Covariance Type: nonrobust
============================================================================================================
coef std err t P>|t| [0.025 0.975]
------------------------------------------------------------------------------------------------------------
Intercept -5898.8935 524.736 -11.242 0.000 -6927.416 -4870.371
C(renovated)[T.True] -0.1557 0.108 -1.436 0.151 -0.368 0.057
C(view)[T.1] 0.1197 0.010 11.587 0.000 0.099 0.140
C(view)[T.2] 0.1110 0.006 17.668 0.000 0.099 0.123
C(view)[T.3] 0.1840 0.009 21.471 0.000 0.167 0.201
C(view)[T.4] 0.2863 0.013 21.680 0.000 0.260 0.312
C(waterfront)[T.1] -39.2234 6.093 -6.437 0.000 -51.167 -27.280
C(zipcode)[T.98002] 0.0198 0.016 1.198 0.231 -0.013 0.052
C(zipcode)[T.98003] -0.0110 0.015 -0.748 0.455 -0.040 0.018
C(zipcode)[T.98004] 0.9112 0.028 32.490 0.000 0.856 0.966
C(zipcode)[T.98005] 0.5227 0.030 17.494 0.000 0.464 0.581
C(zipcode)[T.98006] 0.4607 0.026 17.991 0.000 0.410 0.511
C(zipcode)[T.98007] 0.4495 0.031 14.584 0.000 0.389 0.510
C(zipcode)[T.98008] 0.4584 0.029 15.587 0.000 0.401 0.516
C(zipcode)[T.98010] 0.3090 0.025 12.235 0.000 0.260 0.359
C(zipcode)[T.98011] 0.2708 0.036 7.424 0.000 0.199 0.342
C(zipcode)[T.98014] 0.2036 0.040 5.032 0.000 0.124 0.283
C(zipcode)[T.98019] 0.2236 0.039 5.665 0.000 0.146 0.301
C(zipcode)[T.98022] 0.3401 0.024 13.932 0.000 0.292 0.388
C(zipcode)[T.98023] -0.0619 0.014 -4.574 0.000 -0.088 -0.035
C(zipcode)[T.98024] 0.3202 0.037 8.701 0.000 0.248 0.392
C(zipcode)[T.98027] 0.3814 0.026 14.468 0.000 0.330 0.433
C(zipcode)[T.98028] 0.2104 0.035 5.937 0.000 0.141 0.280
C(zipcode)[T.98029] 0.4810 0.030 16.270 0.000 0.423 0.539
C(zipcode)[T.98030] 0.0044 0.017 0.255 0.798 -0.029 0.038
C(zipcode)[T.98031] -0.0214 0.018 -1.160 0.246 -0.058 0.015
C(zipcode)[T.98032] -0.1272 0.020 -6.262 0.000 -0.167 -0.087
C(zipcode)[T.98033] 0.5770 0.031 18.771 0.000 0.517 0.637
C(zipcode)[T.98034] 0.3307 0.033 10.152 0.000 0.267 0.395
C(zipcode)[T.98038] 0.1937 0.019 10.120 0.000 0.156 0.231
C(zipcode)[T.98039] 1.0794 0.037 29.182 0.000 1.007 1.152
C(zipcode)[T.98040] 0.6642 0.026 25.817 0.000 0.614 0.715
C(zipcode)[T.98042] 0.0530 0.016 3.231 0.001 0.021 0.085
C(zipcode)[T.98045] 0.3211 0.035 9.057 0.000 0.252 0.391
C(zipcode)[T.98052] 0.4592 0.031 14.639 0.000 0.398 0.521
C(zipcode)[T.98053] 0.4574 0.034 13.611 0.000 0.392 0.523
C(zipcode)[T.98055] -0.0020 0.021 -0.095 0.925 -0.044 0.040
C(zipcode)[T.98056] 0.1479 0.023 6.411 0.000 0.103 0.193
C(zipcode)[T.98058] 0.0413 0.020 2.045 0.041 0.002 0.081
C(zipcode)[T.98059] 0.2082 0.023 9.178 0.000 0.164 0.253
C(zipcode)[T.98065] 0.3805 0.033 11.481 0.000 0.316 0.445
C(zipcode)[T.98070] 0.0596 0.025 2.387 0.017 0.011 0.108
C(zipcode)[T.98072] 0.3117 0.036 8.595 0.000 0.241 0.383
C(zipcode)[T.98074] 0.4018 0.031 13.136 0.000 0.342 0.462
C(zipcode)[T.98075] 0.4392 0.030 14.657 0.000 0.381 0.498
C(zipcode)[T.98077] 0.3022 0.038 8.014 0.000 0.228 0.376
C(zipcode)[T.98092] 0.0826 0.015 5.620 0.000 0.054 0.111
C(zipcode)[T.98102] 0.7113 0.032 22.140 0.000 0.648 0.774
C(zipcode)[T.98103] 0.5536 0.030 18.637 0.000 0.495 0.612
C(zipcode)[T.98105] 0.7028 0.031 22.924 0.000 0.643 0.763
C(zipcode)[T.98106] 0.0821 0.024 3.418 0.001 0.035 0.129
C(zipcode)[T.98107] 0.5620 0.031 18.327 0.000 0.502 0.622
C(zipcode)[T.98108] 0.1101 0.026 4.219 0.000 0.059 0.161
C(zipcode)[T.98109] 0.7237 0.032 22.616 0.000 0.661 0.786
C(zipcode)[T.98112] 0.8037 0.029 27.996 0.000 0.747 0.860
C(zipcode)[T.98115] 0.5603 0.030 18.612 0.000 0.501 0.619
C(zipcode)[T.98116] 0.4584 0.026 17.598 0.000 0.407 0.509
C(zipcode)[T.98117] 0.5259 0.030 17.285 0.000 0.466 0.585
C(zipcode)[T.98118] 0.2353 0.024 9.970 0.000 0.189 0.282
C(zipcode)[T.98119] 0.6934 0.030 22.935 0.000 0.634 0.753
C(zipcode)[T.98122] 0.5562 0.028 20.117 0.000 0.502 0.610
C(zipcode)[T.98125] 0.3094 0.032 9.598 0.000 0.246 0.373
C(zipcode)[T.98126] 0.2755 0.024 11.280 0.000 0.228 0.323
C(zipcode)[T.98133] 0.2034 0.033 6.116 0.000 0.138 0.269
C(zipcode)[T.98136] 0.3996 0.025 15.999 0.000 0.351 0.449
C(zipcode)[T.98144] 0.4280 0.026 16.334 0.000 0.377 0.479
C(zipcode)[T.98146] 0.0245 0.023 1.075 0.282 -0.020 0.069
C(zipcode)[T.98148] -0.0269 0.029 -0.924 0.356 -0.084 0.030
C(zipcode)[T.98155] 0.1921 0.035 5.540 0.000 0.124 0.260
C(zipcode)[T.98166] 0.0884 0.021 4.233 0.000 0.047 0.129
C(zipcode)[T.98168] -0.1520 0.022 -6.846 0.000 -0.196 -0.108
C(zipcode)[T.98177] 0.3280 0.035 9.438 0.000 0.260 0.396
C(zipcode)[T.98178] -0.0549 0.023 -2.394 0.017 -0.100 -0.010
C(zipcode)[T.98188] -0.0870 0.023 -3.803 0.000 -0.132 -0.042
C(zipcode)[T.98198] -0.0672 0.017 -3.916 0.000 -0.101 -0.034
C(zipcode)[T.98199] 0.5583 0.029 19.014 0.000 0.501 0.616
np.log(sqft_living) 0.4205 0.006 66.169 0.000 0.408 0.433
np.log(sqft_living):C(renovated)[T.True] 0.0280 0.014 1.972 0.049 0.000 0.056
np.log(sqft_lot) 0.0708 0.002 34.099 0.000 0.067 0.075
bedrooms -0.0141 0.002 -7.791 0.000 -0.018 -0.011
floors 0.0178 0.003 5.492 0.000 0.011 0.024
bathrooms 0.0345 0.003 11.763 0.000 0.029 0.040
condition 0.0451 0.002 20.685 0.000 0.041 0.049
grade 0.1114 0.002 58.335 0.000 0.108 0.115
yr_built -0.0004 7.5e-05 -4.707 0.000 -0.001 -0.000
lat 245.9628 22.097 11.131 0.000 202.652 289.274
lat:C(waterfront)[T.1] 0.8342 0.128 6.508 0.000 0.583 1.085
I(lat ** 2) -2.5821 0.232 -11.106 0.000 -3.038 -2.126
long -0.4103 0.052 -7.876 0.000 -0.512 -0.308
==============================================================================
Omnibus: 1414.225 Durbin-Watson: 2.001
Prob(Omnibus): 0.000 Jarque-Bera (JB): 5898.395
Skew: -0.187 Prob(JB): 0.00
Kurtosis: 5.532 Cond. No. 1.27e+09
==============================================================================
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 1.27e+09. This might indicate that there are
strong multicollinearity or other numerical problems.
Train-Test sets#
df_kc = pd.read_csv(url_base + "kc_house_data.csv")
df_kc = df_kc.set_index("id")
df_kc["renovated"] = df_kc["yr_renovated"].apply(lambda x: 1 if x>0 else 0)
df_kc["log_price"] = np.log(df_kc["price"])
df_kc[["waterfront", "zipcode", "view"]] = df_kc[["waterfront", "zipcode", "view"]].astype("object")
df_kc = df_kc.drop(columns=["sqft_living15", "sqft_lot15", "date", "yr_renovated", "price"])
df_kc.head()
| bedrooms | bathrooms | sqft_living | sqft_lot | floors | waterfront | view | condition | grade | sqft_above | sqft_basement | yr_built | zipcode | lat | long | renovated | log_price | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| id | |||||||||||||||||
| 7129300520 | 3 | 1.00 | 1180 | 5650 | 1.0 | 0 | 0 | 3 | 7 | 1180 | 0 | 1955 | 98178 | 47.5112 | -122.257 | 0 | 12.309982 |
| 6414100192 | 3 | 2.25 | 2570 | 7242 | 2.0 | 0 | 0 | 3 | 7 | 2170 | 400 | 1951 | 98125 | 47.7210 | -122.319 | 1 | 13.195614 |
| 5631500400 | 2 | 1.00 | 770 | 10000 | 1.0 | 0 | 0 | 3 | 6 | 770 | 0 | 1933 | 98028 | 47.7379 | -122.233 | 0 | 12.100712 |
| 2487200875 | 4 | 3.00 | 1960 | 5000 | 1.0 | 0 | 0 | 5 | 7 | 1050 | 910 | 1965 | 98136 | 47.5208 | -122.393 | 0 | 13.311329 |
| 1954400510 | 3 | 2.00 | 1680 | 8080 | 1.0 | 0 | 0 | 3 | 8 | 1680 | 0 | 1987 | 98074 | 47.6168 | -122.045 | 0 | 13.142166 |
X = pd.get_dummies(df_kc, drop_first=True).drop(columns=["log_price"])
y = df_kc["log_price"]
display(X.head())
display(y.head())
| bedrooms | bathrooms | sqft_living | sqft_lot | floors | condition | grade | sqft_above | sqft_basement | yr_built | ... | zipcode_98146 | zipcode_98148 | zipcode_98155 | zipcode_98166 | zipcode_98168 | zipcode_98177 | zipcode_98178 | zipcode_98188 | zipcode_98198 | zipcode_98199 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| id | |||||||||||||||||||||
| 7129300520 | 3 | 1.00 | 1180 | 5650 | 1.0 | 3 | 7 | 1180 | 0 | 1955 | ... | False | False | False | False | False | False | True | False | False | False |
| 6414100192 | 3 | 2.25 | 2570 | 7242 | 2.0 | 3 | 7 | 2170 | 400 | 1951 | ... | False | False | False | False | False | False | False | False | False | False |
| 5631500400 | 2 | 1.00 | 770 | 10000 | 1.0 | 3 | 6 | 770 | 0 | 1933 | ... | False | False | False | False | False | False | False | False | False | False |
| 2487200875 | 4 | 3.00 | 1960 | 5000 | 1.0 | 5 | 7 | 1050 | 910 | 1965 | ... | False | False | False | False | False | False | False | False | False | False |
| 1954400510 | 3 | 2.00 | 1680 | 8080 | 1.0 | 3 | 8 | 1680 | 0 | 1987 | ... | False | False | False | False | False | False | False | False | False | False |
5 rows × 87 columns
id
7129300520 12.309982
6414100192 13.195614
5631500400 12.100712
2487200875 13.311329
1954400510 13.142166
Name: log_price, dtype: float64
# Con sklearn
# encoder = OneHotEncoder(drop="first", sparse_output=False)
# X_categorical_encoded = encoder.fit_transform(df_kc.select_dtypes("object"))
# pd.DataFrame(X_categorical_encoded)
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=0)
X_train
| bedrooms | bathrooms | sqft_living | sqft_lot | floors | condition | grade | sqft_above | sqft_basement | yr_built | ... | zipcode_98146 | zipcode_98148 | zipcode_98155 | zipcode_98166 | zipcode_98168 | zipcode_98177 | zipcode_98178 | zipcode_98188 | zipcode_98198 | zipcode_98199 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| id | |||||||||||||||||||||
| 4045500510 | 1 | 1.00 | 960 | 40946 | 1.0 | 5 | 5 | 960 | 0 | 1945 | ... | False | False | False | False | False | False | False | False | False | False |
| 3824100020 | 3 | 1.75 | 1510 | 9720 | 1.0 | 3 | 7 | 1510 | 0 | 1948 | ... | False | False | False | False | False | False | False | False | False | False |
| 6383000690 | 3 | 2.25 | 1670 | 6414 | 1.0 | 4 | 8 | 1670 | 0 | 1961 | ... | False | False | False | False | False | False | False | False | False | False |
| 2320069014 | 3 | 2.00 | 2660 | 192099 | 1.0 | 4 | 9 | 2660 | 0 | 1964 | ... | False | False | False | False | False | False | False | False | False | False |
| 5315101716 | 3 | 1.75 | 1690 | 13500 | 1.0 | 4 | 7 | 1690 | 0 | 1978 | ... | False | False | False | False | False | False | False | False | False | False |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2322069010 | 5 | 5.00 | 3960 | 94089 | 2.0 | 3 | 10 | 3960 | 0 | 1998 | ... | False | False | False | False | False | False | False | False | False | False |
| 2114700368 | 2 | 2.50 | 1400 | 1262 | 2.0 | 3 | 8 | 1160 | 240 | 2008 | ... | False | False | False | False | False | False | False | False | False | False |
| 5469501200 | 3 | 2.25 | 2360 | 14950 | 1.0 | 4 | 9 | 2360 | 0 | 1978 | ... | False | False | False | False | False | False | False | False | False | False |
| 3751602797 | 4 | 2.00 | 2370 | 76665 | 2.0 | 4 | 8 | 2370 | 0 | 1978 | ... | False | False | False | False | False | False | False | False | False | False |
| 4038600260 | 4 | 2.25 | 2380 | 16236 | 1.0 | 3 | 7 | 1540 | 840 | 1961 | ... | False | False | False | False | False | False | False | False | False | False |
16209 rows × 87 columns
linear_model = LinearRegression()
linear_model.fit(X_train, y_train)
LinearRegression()In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
LinearRegression()
pd.DataFrame(linear_model.coef_, index=X_train.columns, columns=["betas"]).T
| bedrooms | bathrooms | sqft_living | sqft_lot | floors | condition | grade | sqft_above | sqft_basement | yr_built | ... | zipcode_98146 | zipcode_98148 | zipcode_98155 | zipcode_98166 | zipcode_98168 | zipcode_98177 | zipcode_98178 | zipcode_98188 | zipcode_98198 | zipcode_98199 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| betas | 0.004541 | 0.038941 | -4.817226e+06 | 6.605838e-07 | -0.037367 | 0.054626 | 0.104201 | 4.817226e+06 | 4.817226e+06 | -0.000495 | ... | 0.107353 | 0.059306 | 0.17174 | 0.191876 | -0.047642 | 0.335127 | 0.06019 | 0.000487 | -0.004766 | 0.624563 |
1 rows × 87 columns
print(f"Score en train : {linear_model.score(X_train, y_train):.3f}")
print(f"Score en test : {linear_model.score(X_test, y_test):.3f}")
Score en train : 0.875
Score en test : 0.872
Evaluación del rendimiento predictivo#
Para cualquier Dataset, los residuales proporcionan la diferencia entre la predicción del modelo para ese registro y el valor real de ese punto.
El error absoluto medio (MAE) es una métrica que resume el rendimiento holístico (del todo) del modelo en toda la data. El MAE se calcula tomando el valor absoluto de cada residuo y luego tomando la media de todos esos valores absolutos. En esencia, el MAE describe la magnitud típica de los residuales; cuanto menor sea el MAE, mejor.
El error cuadrático medio (RMSE) se calcula tomando el cuadrado de cada residuo, promediando y luego sacando la raíz cuadrada.
Aunque RMSE puede parecer innecesariamente complicado en comparación con MAE, se usa mucho más comúnmente. La razón es que la métrica RMSE es la misma que se minimiza en los datos de entrenamiento mediante un modelo de regresión lineal estándar (también llamado regresión de mínimos cuadrados ordinarios (OLS)). Esto lo convierte en una opción “natural” que utiliza la misma métrica para evaluar el rendimiento fuera de la muestra (test). Considere lo siguiente:
El RMSE pone mucho más peso en los valores atípicos, ya que los errores se elevan al cuadrado antes de promediarlos. En los casos en que los valores atípicos son especialmente malos y deben castigarse, el RMSE es una mejor opción.
Finalmente, está el error porcentual absoluto medio (MAPE). Esto toma el valor absoluto de cada residual y lo divide por el valor real de ese punto para obtener un porcentaje, luego promediando todos los porcentajes.
El MAPE tiene una buena interpretación en la que podemos decir que las predicciones de un modelo son, en promedio, incorrectas en un cierto porcentaje.
# True
y_price_train = np.exp(y_train)
y_price_test = np.exp(y_test)
# Predict
y_price_train_predict = np.exp(linear_model.predict(X_train))
y_price_test_predict = np.exp(linear_model.predict(X_test))
print(f"TRAIN R^2: {r2_score(y_price_train, y_price_train_predict):.4f}")
print(f"TEST R^2: {r2_score(y_price_test, y_price_test_predict):.4f}\n")
print(f"TRAIN MAE: {mean_absolute_error(y_price_train, y_price_train_predict):.4f}")
print(f"TEST MAE: {mean_absolute_error(y_price_test, y_price_test_predict):.4f}\n")
print(f"TRAIN MAPE: {100*mean_absolute_percentage_error(y_price_train, y_price_train_predict):.4f}")
print(f"TEST MAPE: {100*mean_absolute_percentage_error(y_price_test, y_price_test_predict):.4f}")
TRAIN R^2: 0.7184
TEST R^2: 0.7531
TRAIN MAE: 78411.9833
TEST MAE: 76827.7024
TRAIN MAPE: 14.1077
TEST MAPE: 14.0674
df_plot = pd.DataFrame({"valor_verdadero": y_price_train, "valor_predicho": y_price_train_predict})
px.scatter(df_plot, x="valor_verdadero", y="valor_predicho", template="plotly_white")
df_plot = pd.DataFrame({"valor_verdadero": y_price_test, "valor_predicho": y_price_test_predict})
px.scatter(df_plot, x="valor_verdadero", y="valor_predicho", template="plotly_white")
df_review = pd.DataFrame(
{"price": np.exp(y_train), "predict": np.exp(linear_model.predict(X_train))},
index=X_train.index
)
df_review["estimator_diff"] = abs(df_review["price"] - df_review["predict"])
df_review = df_review.sort_values(by=["estimator_diff"], ascending=False).head(100)
df_review = df_kc.merge(df_review, left_index=True, right_index=True)
map = folium.Map(location=[df_kc["lat"].mean(), df_kc["long"].mean()], zoom_start=10, min_zoom=4)
title_html = """
<h2 align="center" style="font-size:20px"><br><b>Mayores diferencia Precios y Predicciones</b></h2>
"""
map.get_root().html.add_child(folium.Element(title_html))
for index, row in df_review.iterrows():
popup_text = f"""
<b>Price:</b> ${int(row['price']):,}<br>
<b>Predicted:</b> ${row['predict']:,.0f}<br>
<b>Difference:</b> ${row['estimator_diff']:,.0f}
<b>Id:</b> {index}
"""
marker = folium.Marker(
location=[row["lat"], row["long"]],
popup=popup_text,
)
icon = folium.Icon(icon="home", prefix="fa", color="green", icon_size=(40, 60))
marker.add_child(icon)
marker.add_to(map)
map
Referencias#
“House Sales in King County, USA”, August 25, 2016, harlfoxem, CC0 Public Domain, https://www.kaggle.com/harlfoxem/housesalesprediction
R. E. Chapman, Degradation Study of a Photographic Developer to Determine Shelf Life, Quality Engineering 10 (1997), pp. 137 – 140.`
