PCA: otros ejemplos y algunas funciones útiles.#
Universidad Central#
Maestría en analítica de datos#
Métodos estadísticos para analítica de datos.#
Docente: Luis Andrés Campos Maldonado.#
Idea central:#
Se tiene un conjunto de \(p\) features cuantitativas y se quieren construir un numero de features \(r\leq p\) con independencia lineal y ortogonales, que contengan la mayor cantidad de variabilidad en el dataset.
Términos clave para el análisis de componentes principales.#
Componente principal: Una combinación lineal de las features originales.
Loading: Los pesos que transforman las features originales en las componentes principales.
Gráfico de sedimentación: Un plot de las varianzas de las componentes, que muestra la importancia relativa de las componentes, ya sea como varianza explicada o como proporción de la varianza explicada.
Representación de Variables Originales en un Biplot de PCA#
El Análisis de Componentes Principales (PCA) es una técnica estadística utilizada para reducir la dimensionalidad de un conjunto de datos, conservando la mayor parte de la varianza. Este proceso implica encontrar nuevas variables (componentes principales) que son combinaciones lineales de las variables originales.
Matriz de Datos Originales#
Supongamos que tenemos un conjunto de datos \(\mathbf{X}\) con \(m\) observaciones y \(n\) variables originales. La matriz \(\mathbf{X}\) tiene la forma:
Antes de realizar el PCA, centramos los datos restando la media de cada variable:
Aquí, \(\mathbf{X}_c\) es la matriz centrada, \(\mathbf{1}\) es un vector columna de unos (de tamaño \(m\)), y \(\mu\) es el vector de medias de cada variable.
Descomposición en Componentes Principales#
Cálculo de la Matriz de Covarianza#
Se calcula la matriz de covarianza \(\mathbf{C}\) de \(\mathbf{X}_c\):
Eigenvalores y Eigenvectores#
Realizamos la descomposición en valores propios de \(\mathbf{C}\):
\(\lambda_i\) son los eigenvalores, que indican la varianza explicada por cada componente principal.
\(\mathbf{v}_i\) son los eigenvectores, que representan las direcciones principales en el espacio original de las \(n\) variables.
Proyección de los Datos#
Las nuevas coordenadas de las observaciones en el espacio de las componentes principales se obtienen proyectando los datos centrados sobre los eigenvectores:
Aquí, \(\mathbf{Z}\) es la matriz de componentes principales, y \(\mathbf{V}_k\) es la matriz de eigenvectores correspondientes a las \(k\) primeras componentes principales.
Representación de las Variables Originales en el Biplot#
Matriz de Cargas#
La matriz \(\mathbf{V}_k\) se conoce como la matriz de cargas. Sus elementos \(v_{ij}\) indican cómo las variables originales contribuyen a las componentes principales:
Cada fila \(\mathbf{v}_j\) representa las cargas de la \(j\)-ésima variable original en las \(k\) componentes principales seleccionadas.
Proyección de Variables en el Espacio de las Componentes Principales#
Para representar las variables originales en un biplot (cuando \(k = 2\)):
Selecciona las columnas de \(\mathbf{V}_k\) correspondientes a las dos primeras componentes principales, \(\mathbf{v}_1\) y \(\mathbf{v}_2\).
La proyección de la \(j\)-ésima variable original en el biplot se representa por el punto \((v_{j1}, v_{j2})\), donde:
\(v_{j1}\) es la carga de la \(j\)-ésima variable en la primera componente principal.
\(v_{j2}\) es la carga de la \(j\)-ésima variable en la segunda componente principal.
Visualización en el Biplot#
Para cada variable \(j\), se dibuja un vector desde el origen hasta el punto \((v_{j1}, v_{j2})\). Estos vectores en el biplot indican:
Dirección: Hacia dónde “apunta” la variable en relación con las componentes principales.
Longitud: Cuánta varianza de la variable original es capturada por las dos primeras componentes principales.
Interpretación#
Ángulo entre vectores: Indica la correlación entre las variables originales.
Longitud del vector: Indica la contribución de la variable a las componentes principales seleccionadas.
Dirección: Muestra cómo se relaciona cada variable con las nuevas dimensiones definidas por las componentes principales.
Resumen#
En un biplot, los vectores que representan las variables originales son el resultado de proyectar estas variables en el espacio definido por las componentes principales seleccionadas. Esta proyección se realiza utilizando la matriz de cargas, y los vectores resultantes permiten visualizar e interpretar cómo las variables originales contribuyen a la estructura de varianza capturada en el espacio de las componentes principales.
Protocolo de módulos PCA.#
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import plotly.express as px
import plotly.graph_objects as go
import warnings
##
from sklearn.decomposition import PCA ## Principal component analysis.
from sklearn.preprocessing import StandardScaler ## Escalar datos.
from utils.plots_pca import *
##
warnings.filterwarnings('ignore')
plt.rcParams['figure.figsize'] = (15, 9)
plt.style.use('ggplot')
Construcción de algunas funciones auxiliares#
En los siguientes bloques de código se muestran algunas funciones que pueden de ser de ayuda para el ánalisis de PCA en Python.
Todas fueron construidas por el autor de este cuaderno, con apoyo del material de la página oficial de plotly, tenga la libertad de entenderla, mejorarla y por supuesto compartir dicha mejoras.
biplotEsta función genera un biplot donde se muestran los espacios de individuos y las variables en un solo plot.biplot1: Esta función genera solo el primer plano factorial donde se aprecia la influencia de la features originales en dicho plano,view_index: Permite seleccionar algunos registros del dataframe en pro de un mejor entendimiento de los plots anteriores.sedimentacion: Genera el gráfico de sedimentación para buscar un posible método para la selección de la cantidad de componenetes principales.plot_loadings_heatmap(): Genera un barplot de las cargas en cada una de las componentes principales.plot_loadings_bar(): Esta función genera un heatmap de los loadings de las componentes pricipales.
Ejemplo 1#
El dataset birds.txt contiene medidas corporales de aves, se desea buscar realizar un análisis de la selección natural.
Se tiene una información adicional sobre el índice de los registros, de la ave 1 a la 21 sobrevivieron.
url_base = "https://raw.githubusercontent.com/lacamposm/Metodos_Estadisticos/main/data/"
df_name1 = "birds.txt"
df1 = pd.read_csv(url_base + df_name1, sep = " ", index_col = 0,)
df1.head()
| longitud_total | extension_alas | longitud_pico_y_cabeza | longitud_humero | longitud_quilla | |
|---|---|---|---|---|---|
| pajaro | |||||
| 1 | 156 | 245 | 31.6 | 18.5 | 20.5 |
| 2 | 154 | 240 | 30.4 | 17.9 | 19.6 |
| 3 | 153 | 240 | 31.0 | 18.4 | 20.6 |
| 4 | 153 | 236 | 30.9 | 17.7 | 20.2 |
| 5 | 155 | 243 | 31.5 | 18.6 | 20.3 |
sns.heatmap(df1.corr(), annot=True, cmap="PuBu")
plt.show()
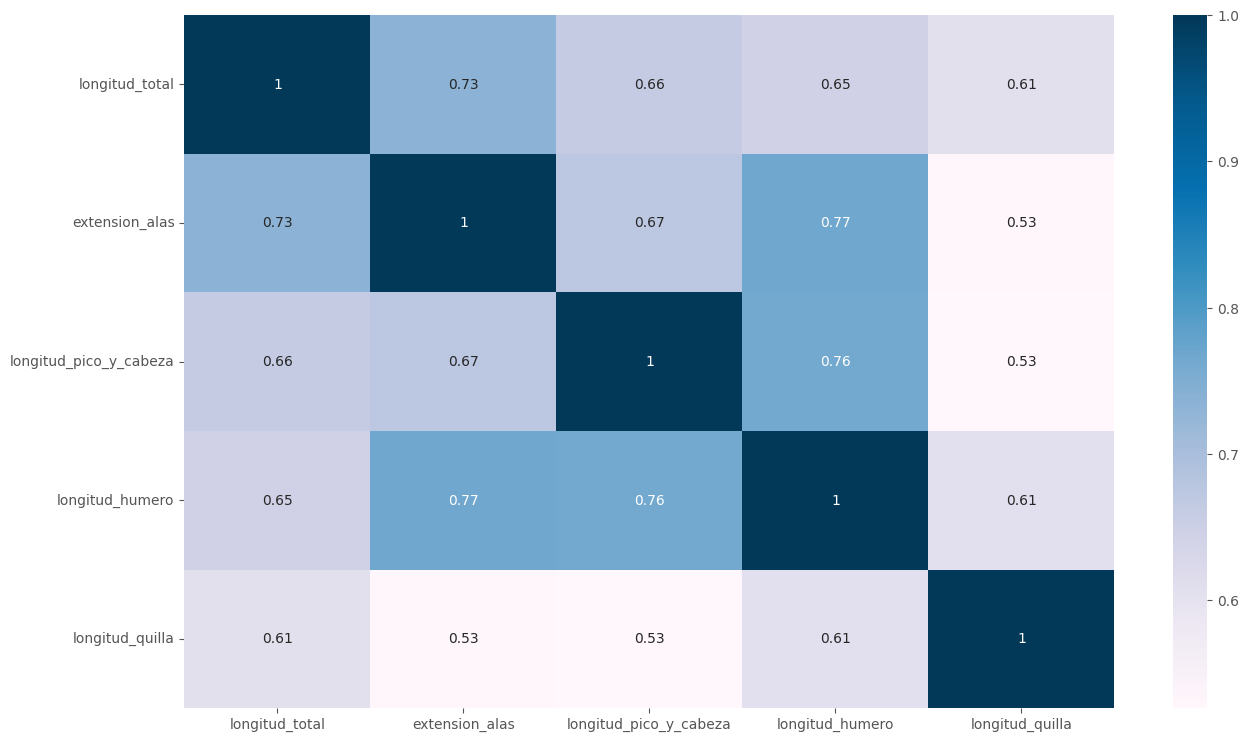
Podemos observar una alta correlación lineal entre las variables, podemos pensar que un PCA podría estar bastaste bien en este caso.
# Rutina habitual.
scaler1 = StandardScaler()
scaler1.fit(df1)
X_scaled1 = scaler1.transform(df1)
display(pd.DataFrame(X_scaled1, columns=df1.columns))
pca1 = PCA()
pca1.fit(X_scaled1)
| longitud_total | extension_alas | longitud_pico_y_cabeza | longitud_humero | longitud_quilla | |
|---|---|---|---|---|---|
| 0 | -0.547333 | 0.732373 | 0.179019 | 0.054812 | -0.332785 |
| 1 | -1.100309 | -0.264468 | -1.346531 | -1.019498 | -1.250023 |
| 2 | -1.376796 | -0.264468 | -0.583756 | -0.124240 | -0.230870 |
| 3 | -1.376796 | -1.061941 | -0.710885 | -1.377602 | -0.638531 |
| 4 | -0.823821 | 0.333637 | 0.051889 | 0.233863 | -0.536616 |
| 5 | 1.388081 | 1.131110 | 0.687535 | 0.950070 | 0.074877 |
| 6 | -0.270845 | -0.663205 | -0.710885 | -0.124240 | -0.638531 |
| 7 | -0.823821 | -0.463836 | 1.704568 | 0.233863 | 0.380623 |
| 8 | 1.664569 | 1.330478 | 1.577439 | 1.129122 | 0.278707 |
| 9 | 0.005643 | -0.663205 | -0.583756 | 0.591967 | 1.195946 |
| 10 | 0.005643 | -0.264468 | -0.202369 | 0.233863 | 1.195946 |
| 11 | 0.558618 | 0.533005 | -0.456627 | 0.233863 | -0.332785 |
| 12 | 0.835106 | 0.931742 | 1.068923 | 1.487225 | 0.992115 |
| 13 | -0.270845 | 0.732373 | 0.687535 | 1.129122 | -0.842362 |
| 14 | -0.270845 | -1.261309 | 0.051889 | -0.661395 | -1.046193 |
| 15 | -0.547333 | -0.862573 | -0.710885 | -0.840447 | -0.536616 |
| 16 | 0.005643 | 0.533005 | -0.075240 | 0.054812 | 0.788284 |
| 17 | -1.376796 | -0.663205 | -1.219402 | -0.482343 | 0.074877 |
| 18 | -0.823821 | -1.061941 | -1.473660 | 0.054812 | -0.740446 |
| 19 | 1.388081 | 0.931742 | 1.323181 | 0.233863 | 1.094030 |
| 20 | 0.282130 | -1.061941 | 0.051889 | -0.840447 | 0.686369 |
| 21 | -0.823821 | -0.264468 | -0.075240 | -0.840447 | -0.128954 |
| 22 | -0.547333 | -0.264468 | 0.051889 | -0.482343 | -0.230870 |
| 23 | 0.558618 | 0.134268 | 1.450310 | 0.591967 | 0.890200 |
| 24 | -1.653284 | -1.859414 | -1.473660 | -2.272860 | -1.046193 |
| 25 | 0.558618 | 1.729215 | 0.306148 | 0.591967 | 1.705523 |
| 26 | -0.823821 | -0.862573 | -0.583756 | 0.054812 | -0.842362 |
| 27 | -0.270845 | 0.732373 | 0.941793 | 1.845328 | 0.584454 |
| 28 | 1.941057 | 0.732373 | 2.085956 | 2.382483 | 1.909353 |
| 29 | -1.376796 | -2.058783 | -1.727919 | -2.093808 | -1.046193 |
| 30 | 1.111594 | -0.463836 | -1.473660 | -0.840447 | 2.317015 |
| 31 | 1.111594 | 0.333637 | 0.179019 | 0.591967 | 0.482538 |
| 32 | 0.282130 | 0.732373 | 0.433277 | 0.054812 | 0.890200 |
| 33 | 0.282130 | 1.131110 | -0.710885 | -0.661395 | -1.861516 |
| 34 | -0.823821 | 0.333637 | -0.710885 | 0.054812 | 0.482538 |
| 35 | 1.111594 | 2.127951 | 0.560406 | 1.129122 | 1.399777 |
| 36 | -1.653284 | -2.258151 | -1.346531 | -2.093808 | -2.269177 |
| 37 | 0.282130 | 0.134268 | -0.838015 | -0.482343 | -0.332785 |
| 38 | -0.823821 | -0.663205 | -0.329498 | -1.019498 | -1.555770 |
| 39 | 1.388081 | 1.529846 | 2.467343 | 1.845328 | 2.011269 |
| 40 | 1.388081 | 0.134268 | -0.583756 | -0.661395 | -0.128954 |
| 41 | -0.547333 | -0.862573 | 0.306148 | -0.482343 | -0.536616 |
| 42 | 0.282130 | -0.663205 | 0.051889 | -0.124240 | -0.536616 |
| 43 | 0.835106 | 0.732373 | 0.814664 | 1.129122 | -0.027039 |
| 44 | -0.823821 | -1.261309 | -0.965144 | -1.377602 | -1.250023 |
| 45 | 1.111594 | 1.131110 | 0.560406 | 1.129122 | -0.434700 |
| 46 | -1.376796 | -0.862573 | -1.092273 | 0.233863 | -0.434700 |
| 47 | 1.111594 | 0.732373 | 1.323181 | 0.054812 | 0.278707 |
| 48 | 1.664569 | 1.330478 | 1.068923 | 0.591967 | 0.074877 |
PCA()In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
PCA()
# Plot de sediementación
sedimentacion(pca1)
Notemos la forma de codo que se presenta en la gráfica, dado que no hay un método puntual para la selección de las componentes principal, podemos optar por incluir cierta cantidad de varibilidad (un valor determinado) o determinar el número de componentes principales observando si hasta determinado punto la inclusión de una variable adicional tiene un peso significativo, recuerde que a más variables la interpretabilidad se vuelve bastante complicada.
Basados en lo anterior vamos a considerar vamos a conciderar solo 2 componentes principales.
print(pca1.explained_variance_)
print(pca1.explained_variance_ratio_)
print(np.cumsum(pca1.explained_variance_ratio_))
[3.69131123 0.54257708 0.39447506 0.30784813 0.16795517]
[0.72319567 0.10630082 0.07728491 0.0603131 0.0329055 ]
[0.72319567 0.82949648 0.90678139 0.9670945 1. ]
n_components = 2
pca1 = PCA(n_components=n_components)
pca1.fit(X_scaled1)
PCA(n_components=2)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
PCA(n_components=2)
expl = pca1.explained_variance_ratio_*100
print("""Con las {} primeras componentes principales, la cantidad de varianza explicada es: {:.2f}%
""".format(n_components,sum(expl[0: n_components])))
Con las 2 primeras componentes principales, la cantidad de varianza explicada es: 82.95%
# Loadings.
plot_loadings_heatmap(pca1, df1)
Recuerde que los valores que se generan apartir de la primera componente principal es una combinación lineal de la forma:
Vemos que todos los coeficientes son positivos, y además, aproximadamente el \(72.3\%\) de la variación está relacionada con las diferencias de tamaño, luego \(Y_1\) es un indicador del tamaño de las aves.
De manera completamente análoga, \(Y_2\), reúne las variables que registran la forma de las aves, pues se contrastan las features: extensión de las alas, longitud de pico y cabeza, extensión del húmero con longitud de la quilla. Mientras el primer grupo aumenta, la longitud de la quilla disminuye y viceversa.
La información transformada se encuentra es el siguiente código.
pca1_trans = pca1.transform(X_scaled1)
pca1_trans = pd.DataFrame(pca1_trans, index=df1.index, columns=["PC1","PC2"])
pca1_trans.head()
| PC1 | PC2 | |
|---|---|---|
| pajaro | ||
| 1 | 0.064955 | -0.607064 |
| 2 | -2.202907 | -0.446884 |
| 3 | -1.157437 | 0.019454 |
| 4 | -2.335015 | 0.173775 |
| 5 | -0.298100 | -0.672101 |
print("Se ajustó un PCA a una data de tamaño:",df1.shape)
print("Se obtuvo un ajuste por PCA de tamaño:",pca1_trans.shape)
Se ajustó un PCA a una data de tamaño: (49, 5)
Se obtuvo un ajuste por PCA de tamaño: (49, 2)
biplot(pca1,df1)
Teniendo en cuenta lo comentado en el párrafo anterior, la ave número 31 (arriba en el centro) tiene el valor más alto respecto a la longitud de la quilla. Las aves 30, 25, 37 (a la izquierda) tienen valores bajos (con respecto a la medía) en varias features (muchas ponderan en el primer componente principal). En constraste tenemos las aves 29, 40. La ave “propotípo” esta en el centro del plot (ave 41).
df1.mean()
longitud_total 157.979592
extension_alas 241.326531
longitud_pico_y_cabeza 31.459184
longitud_humero 18.469388
longitud_quilla 20.826531
dtype: float64
view_index(df1, [31,30,25,37,29,40])
| longitud_total | extension_alas | longitud_pico_y_cabeza | longitud_humero | longitud_quilla | |
|---|---|---|---|---|---|
| 31 | 162.0 | 239.0 | 30.3 | 18.0 | 23.1 |
| 30 | 153.0 | 231.0 | 30.1 | 17.3 | 19.8 |
| 25 | 152.0 | 232.0 | 30.3 | 17.2 | 19.8 |
| 37 | 152.0 | 230.0 | 30.4 | 17.3 | 18.6 |
| 29 | 165.0 | 245.0 | 33.1 | 19.8 | 22.7 |
| 40 | 163.0 | 249.0 | 33.4 | 19.5 | 22.8 |
El siguiente plot:
biplot1(pca1, df1)
se puede observar como el primer eje en el plano factorial está influenciado por la longitud del húmero y la longitud total, para el segundo eje factorial, longitud de la quilla y longitud del pico y cabeza.
## Selección natural.
temp = pca1_trans.copy()
temp["sobrevivio"] = ["vivo" if x<22 else "muerto" for x in df1.index.values]
px.scatter(
temp,
x="PC1",
y="PC2",
color="sobrevivio"
)
Ejercicio en clase.#
Realizar un PCA con la base de emisiones y teniendo como individuo el año. Realice toda la rutina presentada en clase, busque generar sus propios análisis haciendo uso de las nuevas herramientas presentados en este cuaderno.
Retomando un ejemplo anterior.#
Consideremos el dataset Global_Carbon_Budget_2018.csv.
url_base = "https://raw.githubusercontent.com/lacamposm/Metodos_Estadisticos/main/data/"
df_name = "Global_Carbon_Budget_2018.csv"
df = pd.read_csv(url_base + df_name, sep=";", decimal=",", index_col = "Year")
df.head()
| fossil fuel and industry | land-use change emissions | atmospheric growth | ocean sink | land sink | budget imbalance | |
|---|---|---|---|---|---|---|
| Year | ||||||
| 1750 | NaN | NaN | -0.08 | NaN | 0.09 | NaN |
| 1751 | 0.0 | NaN | -0.07 | NaN | -0.53 | NaN |
| 1752 | 0.0 | NaN | -0.07 | NaN | -0.27 | NaN |
| 1753 | 0.0 | NaN | -0.07 | NaN | -0.17 | NaN |
| 1754 | 0.0 | NaN | -0.06 | NaN | -0.33 | NaN |
feature |
Traducción |
|---|---|
fossil fuel and industry |
Combustible fósil e industría |
land-use change emissions |
emisiones de cambio de uso de la tierra |
atmospheric growth |
crecimiento atmosférico |
ocean sink |
sumidero del oceano |
land sink |
sumidero de tierra |
budget imbalance |
desequilibrio presupuestario |
## Rutina escalamiento.
df_pca = df.drop(columns=["budget imbalance"])
df_pca = df_pca.dropna()
scaler = StandardScaler()
scaler.fit(df_pca)
X_scaled = scaler.transform(df_pca)
pd.DataFrame(X_scaled, index=df_pca.index, columns=df_pca.columns).corr()
| fossil fuel and industry | land-use change emissions | atmospheric growth | ocean sink | land sink | |
|---|---|---|---|---|---|
| fossil fuel and industry | 1.000000 | 0.404771 | 0.969705 | 0.988774 | 0.727075 |
| land-use change emissions | 0.404771 | 1.000000 | 0.293668 | 0.426617 | 0.295279 |
| atmospheric growth | 0.969705 | 0.293668 | 1.000000 | 0.966279 | 0.711759 |
| ocean sink | 0.988774 | 0.426617 | 0.966279 | 1.000000 | 0.726006 |
| land sink | 0.727075 | 0.295279 | 0.711759 | 0.726006 | 1.000000 |
# Rutina PCA
pca = PCA()
pca.fit(X_scaled)
pca_array = pca.transform(X_scaled)
# Plot de sedimentación
sedimentacion(pca)
name_columns = [f"PC{i+1}" for i in range(0,pca_array.shape[1])]
pca_trans = pd.DataFrame(pca_array, index = df_pca.index, columns = name_columns)
pca_trans.head()
| PC1 | PC2 | PC3 | PC4 | PC5 | |
|---|---|---|---|---|---|
| Year | |||||
| 1850 | -2.095784 | -1.196339 | 0.250146 | -0.083911 | 0.128713 |
| 1851 | -2.381818 | -1.092507 | -0.303762 | -0.142315 | 0.118502 |
| 1852 | -2.263906 | -1.095204 | -0.005834 | -0.179125 | 0.122529 |
| 1853 | -2.378335 | -1.053040 | -0.175729 | -0.224897 | 0.123368 |
| 1854 | -2.352181 | -1.059783 | -0.066733 | -0.261231 | 0.126965 |
pca.components_
array([[ 0.50625924, 0.25177241, 0.49391515, 0.50715776, 0.42326242],
[-0.08557012, 0.95984491, -0.2149806 , -0.0587595 , -0.14733009],
[-0.24669646, 0.03728889, -0.28105587, -0.24452182, 0.89384958],
[-0.41578379, 0.11667435, 0.79418177, -0.42733142, 0.01319523],
[ 0.70897393, 0.01746506, -0.01068107, -0.70493636, -0.0012577 ]])
# Plot loadings.
plot_loadings_heatmap(pca,df_pca,n_compo=5)
plot_loadings_bar(pca, df_pca, n_compo=5, rotation=40)
biplot(pca, df_pca)
df_pca.mean()
fossil fuel and industry 1.999677
land-use change emissions 1.182645
atmospheric growth 1.204452
ocean sink 0.834645
land sink 1.008129
dtype: float64
view_index(df_pca,[1867,1958,1997,1999])
| fossil fuel and industry | land-use change emissions | atmospheric growth | ocean sink | land sink | |
|---|---|---|---|---|---|
| 1867 | 0.13 | 0.67 | 0.39 | 0.25 | 0.55 |
| 1958 | 2.33 | 1.79 | 1.02 | 0.81 | -0.06 |
| 1997 | 6.53 | 1.78 | 3.39 | 2.14 | 2.98 |
| 1999 | 6.53 | 1.20 | 3.43 | 2.28 | 3.70 |
biplot1(pca,df_pca)
Ejemplo 2.#
Vamos a considerar el dataframe Delitos_Colombia.csv que contiene el número de delitos por departamento, en distintas categorías. Toda la información es numérica.
url_base = "https://raw.githubusercontent.com/lacamposm/Metodos_Estadisticos/main/data/"
df_name2 = "Delitos_Colombia.csv"
df2 = pd.read_csv(url_base + df_name2, sep=";", decimal=",", index_col=0)
df2.head()
| Delitos_Sexuales | Homicidios | Transito | Asalto | Intrafamiliar | Poblacion | |
|---|---|---|---|---|---|---|
| Departamento | ||||||
| Antioquia | 2163 | 375 | 5079 | 11897 | 8205 | 6690977 |
| Atlantico | 1042 | 85 | 1928 | 59 | 3659 | 2546138 |
| Bogota_D.C. | 4211 | 1463 | 725 | 2725 | 19811 | 8181047 |
| Bolivar | 944 | 28 | 922 | 3812 | 2085 | 2171558 |
| Boyaca | 517 | 95 | 1167 | 4084 | 2707 | 1281979 |
df2.describe().T
| count | mean | std | min | 25% | 50% | 75% | max | |
|---|---|---|---|---|---|---|---|---|
| Delitos_Sexuales | 33.0 | 6.484545e+02 | 8.246769e+02 | 0.0 | 204.0 | 420.0 | 616.0 | 4211.0 |
| Homicidios | 33.0 | 1.161818e+02 | 2.596099e+02 | 0.0 | 17.0 | 58.0 | 81.0 | 1463.0 |
| Transito | 33.0 | 1.173667e+03 | 1.371468e+03 | 0.0 | 143.0 | 861.0 | 1248.0 | 5599.0 |
| Asalto | 33.0 | 2.816121e+03 | 3.001322e+03 | 1.0 | 654.0 | 2243.0 | 3766.0 | 11897.0 |
| Intrafamiliar | 33.0 | 2.059242e+03 | 3.590915e+03 | 2.0 | 411.0 | 1083.0 | 2142.0 | 19811.0 |
| Poblacion | 33.0 | 1.510143e+06 | 1.827319e+06 | 43446.0 | 375258.0 | 1040193.0 | 1788648.0 | 8181047.0 |
# Seleccionamos las cuantitativas (ACP solo trabaja con cuantitativas). Cuando sea el caso seleccionamos
# así: -----> cuanti = df2.select_dtypes(np.number)
sns.heatmap(df2.corr(), annot = True, cmap = "PuBu")
plt.show()
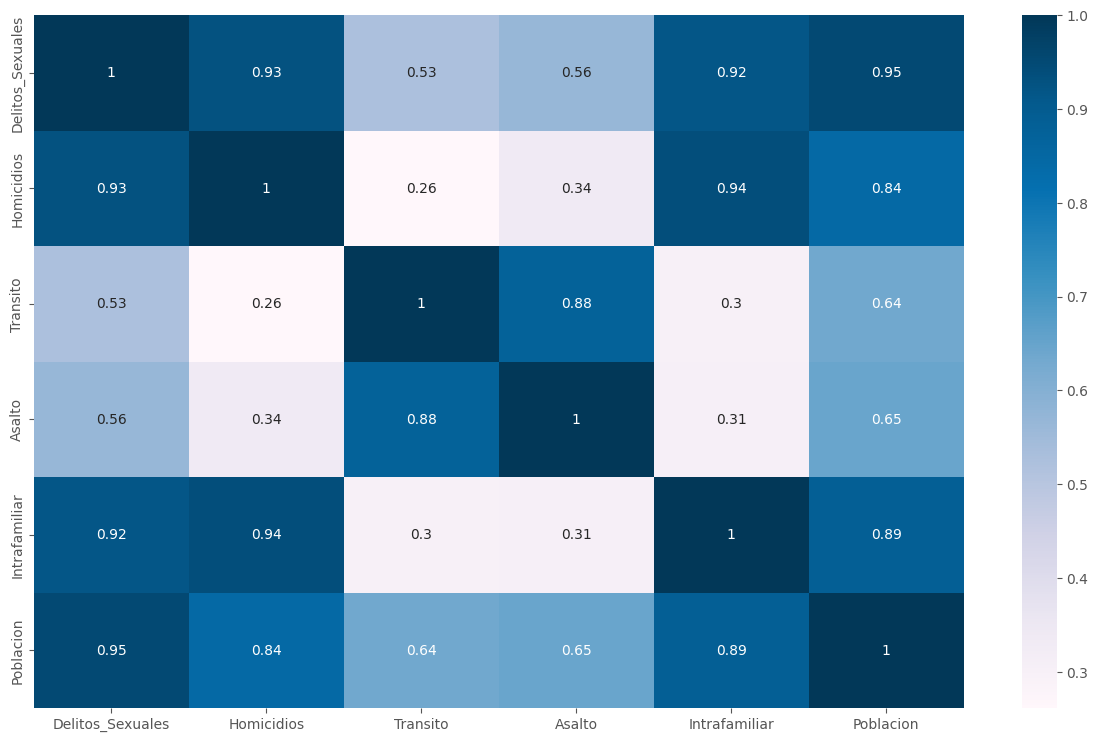
Notemos que las diferencias en las desviaciones en las distintas features es bastante alta, debemos por tanto realizar unas “mejoras” en nuestro dataset.
Para realizar un mejor análisis vamos considerar la siguiente tasa (para todas las variables):
la cual representa la incidencia por cada \(100.000\) habitantes.
df2_pca = df2.copy()
df2_pca["DSex_p"] = 100000*df2_pca["Delitos_Sexuales"]/df2_pca["Poblacion"]
df2_pca["DHom_p"] = 100000*df2_pca["Homicidios"]/df2_pca["Poblacion"]
df2_pca["DIntra_p"] = 100000*df2_pca["Intrafamiliar"]/df2_pca["Poblacion"]
df2_pca["DTransi_p"] = 100000*df2_pca["Transito"]/df2_pca["Poblacion"]
df2_pca["DAsal_p"] = 100000*df2_pca["Asalto"]/df2_pca["Poblacion"]
df2_pca = df2_pca[["DSex_p", "DHom_p", "DIntra_p", "DTransi_p", "DAsal_p"]]
df2_pca
| DSex_p | DHom_p | DIntra_p | DTransi_p | DAsal_p | |
|---|---|---|---|---|---|
| Departamento | |||||
| Antioquia | 32.327118 | 5.604563 | 122.627831 | 75.908197 | 177.806619 |
| Atlantico | 40.924726 | 3.338389 | 143.707843 | 75.722526 | 2.317235 |
| Bogota_D.C. | 51.472629 | 17.882797 | 242.157269 | 8.861946 | 33.308695 |
| Bolivar | 43.471093 | 1.289397 | 96.014014 | 42.457996 | 175.542168 |
| Boyaca | 40.328274 | 7.410418 | 211.157905 | 91.031132 | 318.569961 |
| Caldas | 44.573234 | 6.540091 | 98.101361 | 125.569743 | 225.683439 |
| Caqueta | 44.532928 | 3.627116 | 90.476402 | 24.382282 | 131.785226 |
| Cauca | 29.657980 | 4.378083 | 104.156001 | 60.798859 | 167.920658 |
| Cesar | 52.644568 | 4.128986 | 131.564501 | 79.013773 | 258.718494 |
| Cordoba | 29.631319 | 0.782714 | 36.172573 | 49.255080 | 95.938385 |
| Cundinamarca | 52.028394 | 12.873372 | 22.359015 | 91.611340 | 367.051584 |
| Choco | 30.863838 | 0.388224 | 56.874871 | 19.605331 | 102.491236 |
| Huila | 41.602307 | 5.680636 | 184.286525 | 92.143262 | 318.700404 |
| La_Guajira | 30.667386 | 2.595672 | 72.390412 | 18.650385 | 119.593191 |
| Magdalena | 38.504130 | 1.925206 | 141.849215 | 53.135699 | 230.485722 |
| Meta | 81.245475 | 9.245853 | 272.359227 | 108.491234 | 370.424286 |
| narigno | 10.998723 | 4.255787 | 82.960215 | 98.988504 | 199.248218 |
| Norte_de_Santander | 26.880059 | 4.887283 | 156.968044 | 76.830970 | 235.883297 |
| Quindio | 69.570057 | 14.087937 | 169.750939 | 211.666899 | 354.633366 |
| Risaralda | 55.797805 | 7.543037 | 144.247660 | 222.054599 | 232.594185 |
| Santander | 62.127724 | 8.943714 | 19.035284 | 156.730216 | 357.461592 |
| Sucre | 46.064874 | 1.938373 | 123.485788 | 30.899953 | 192.469077 |
| Tolima | 43.381595 | 4.788877 | 150.849638 | 186.132397 | 300.220359 |
| Valle_del_Cauca | 39.341767 | 5.950679 | 108.205629 | 117.730920 | 198.832574 |
| Arauca | 82.745985 | 15.884274 | 316.577271 | 79.421369 | 291.827356 |
| Casanare | 69.285665 | 15.456033 | 344.562941 | 160.422962 | 368.812923 |
| Putumayo | 56.840979 | 6.129910 | 114.517855 | 39.844412 | 194.485311 |
| San_Andres | 29.331871 | 5.101195 | 300.970502 | 137.732264 | 629.997577 |
| Amazonas | 83.724470 | 3.805658 | 258.784727 | 30.445262 | 318.406698 |
| Guainia | 85.163191 | 39.129034 | 135.800764 | 64.447820 | 202.550292 |
| Guaviare | 49.210474 | 5.180050 | 113.961098 | 18.130175 | 123.457856 |
| Vaupes | 0.000000 | 0.000000 | 4.451567 | 0.000000 | 2.225783 |
| Vichada | 15.528754 | 1.294063 | 38.821885 | 51.762514 | 78.937833 |
sns.heatmap(df2_pca.corr(), annot = True, cmap = "PuBu")
plt.show()
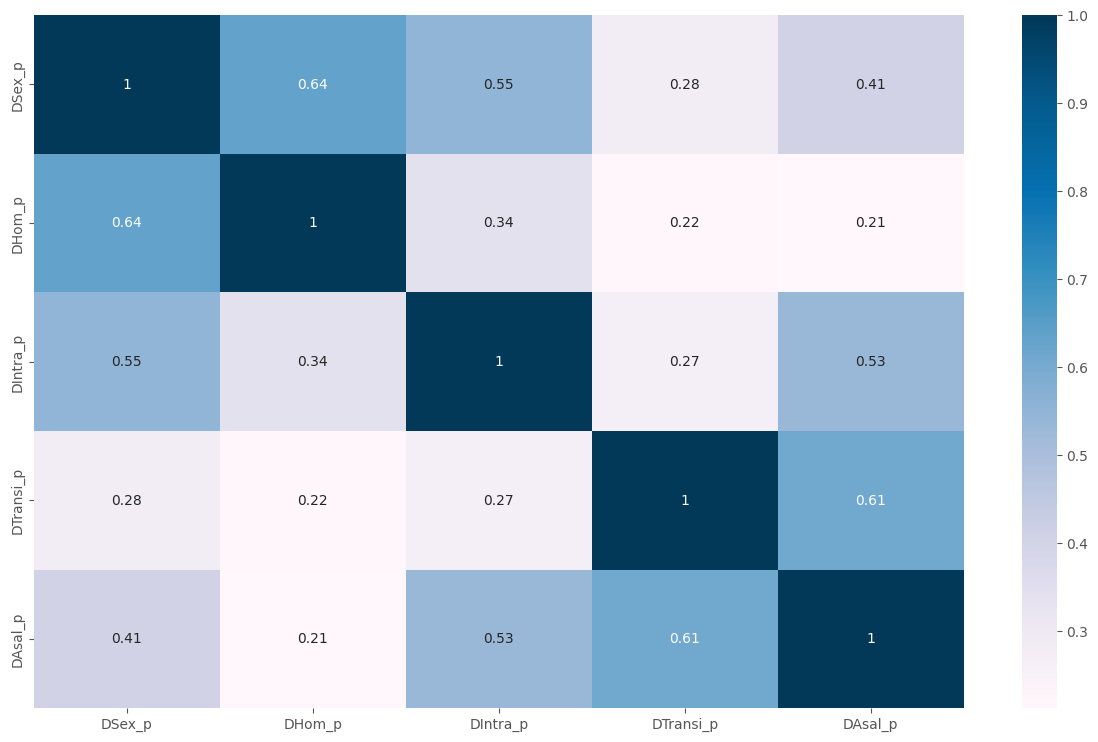
Vamos escalar la información para buscar mitigar la variables que tienen altas varianzas, ya que estas se llevarian el mayor de la variabilidad total. Deseamos que su variabilidad sea mitigada.
scaler2 = StandardScaler() # Cargando el escalador estandar
scaler2.fit(df2_pca) # Calcula las medias y las desviaciones
X_scaled = scaler2.transform(df2_pca) # Estandariza la data.
pd.DataFrame(X_scaled, index = df2_pca.index, columns = df2_pca.columns)
| DSex_p | DHom_p | DIntra_p | DTransi_p | DAsal_p | |
|---|---|---|---|---|---|
| Departamento | |||||
| Antioquia | -0.663883 | -0.196509 | -0.199243 | -0.104096 | -0.361624 |
| Atlantico | -0.239343 | -0.508407 | 0.046727 | -0.107368 | -1.747823 |
| Bogota_D.C. | 0.281500 | 1.493371 | 1.195478 | -1.285749 | -1.503020 |
| Bolivar | -0.113606 | -0.790414 | -0.509785 | -0.693637 | -0.379511 |
| Boyaca | -0.268795 | 0.052035 | 0.833764 | 0.162437 | 0.750273 |
| Caldas | -0.059184 | -0.067750 | -0.485429 | 0.771161 | 0.016557 |
| Caqueta | -0.061174 | -0.468669 | -0.574400 | -1.012212 | -0.725149 |
| Cauca | -0.795682 | -0.365312 | -0.414780 | -0.370390 | -0.439714 |
| Cesar | 0.339369 | -0.399596 | -0.094966 | -0.049362 | 0.277503 |
| Cordoba | -0.796998 | -0.860150 | -1.208040 | -0.573843 | -1.008305 |
| Cundinamarca | 0.308943 | 0.803913 | -1.369223 | 0.172663 | 1.133231 |
| Choco | -0.736138 | -0.914445 | -0.966477 | -1.096403 | -0.956544 |
| Huila | -0.205885 | -0.186039 | 0.520217 | 0.182038 | 0.751303 |
| La_Guajira | -0.745839 | -0.610629 | -0.785435 | -1.113233 | -0.821455 |
| Magdalena | -0.358869 | -0.702906 | 0.025040 | -0.505449 | 0.054491 |
| Meta | 1.751649 | 0.304650 | 1.547887 | 0.470162 | 1.159873 |
| narigno | -1.717055 | -0.382144 | -0.662102 | 0.302682 | -0.192256 |
| Norte_de_Santander | -0.932852 | -0.295230 | 0.201453 | -0.087833 | 0.097127 |
| Quindio | 1.175131 | 0.971076 | 0.350610 | 2.288576 | 1.035139 |
| Risaralda | 0.495073 | 0.070287 | 0.053026 | 2.471654 | 0.071146 |
| Santander | 0.807637 | 0.263066 | -1.408006 | 1.320347 | 1.057480 |
| Sucre | 0.014472 | -0.701094 | -0.189232 | -0.897342 | -0.245805 |
| Tolima | -0.118026 | -0.308773 | 0.130061 | 1.838545 | 0.605328 |
| Valle_del_Cauca | -0.317508 | -0.148872 | -0.367528 | 0.633006 | -0.195539 |
| Arauca | 1.825743 | 1.218310 | 2.063842 | -0.042178 | 0.539031 |
| Casanare | 1.161088 | 1.159370 | 2.390391 | 1.385430 | 1.147144 |
| Putumayo | 0.546583 | -0.124204 | -0.293874 | -0.739700 | -0.229878 |
| San_Andres | -0.811785 | -0.265788 | 1.881736 | 0.985519 | 3.210255 |
| Amazonas | 1.874059 | -0.444096 | 1.389494 | -0.905355 | 0.748983 |
| Guainia | 1.945102 | 4.417535 | -0.045536 | -0.306079 | -0.166173 |
| Guaviare | 0.169798 | -0.254935 | -0.300370 | -1.122402 | -0.790928 |
| Vaupes | -2.260159 | -0.967877 | -1.578175 | -1.441936 | -1.748546 |
| Vichada | -1.493367 | -0.789772 | -1.177127 | -0.529650 | -1.142594 |
# Realizamos la rutina habitual.
pca2 = PCA(n_components = 0.85)
pca2.fit(X_scaled)
n_comp, var_exp = len(pca2.explained_variance_ratio_), sum(pca2.explained_variance_ratio_)
print("Con {} componentes se explica el {:.2f}% de la variabilidad".format(n_comp,var_exp*100))
Con 3 componentes se explica el 87.92% de la variabilidad
# Plot de sedimentación
sedimentacion(pca2)
pca2_trans = pca2.transform(X_scaled)
pca2_trans = pd.DataFrame(pca2_trans, index = df2.index, columns = ["PC1","PC2","PC3"])
pca2_trans.head()
| PC1 | PC2 | PC3 | |
|---|---|---|---|
| Departamento | |||
| Antioquia | -0.712187 | 0.151438 | -0.039164 |
| Atlantico | -1.168391 | -0.511234 | 0.088261 |
| Bogota_D.C. | 0.086747 | -2.423493 | 0.615291 |
| Bolivar | -1.062042 | -0.059052 | 0.315585 |
| Boyaca | 0.694155 | 0.482570 | 0.583906 |
print("Se ajustó un PCA a una data de tamaño:",df2_pca.shape)
print("Se obtuvo un ajuste por PCA de tamaño:",pca2_trans.shape)
expl = pca2.explained_variance_ratio_
print("Cantidad de varianza explicada: {:.2f}%".format(sum(expl[0:3])*100))
Se ajustó un PCA a una data de tamaño: (33, 5)
Se obtuvo un ajuste por PCA de tamaño: (33, 3)
Cantidad de varianza explicada: 87.92%
# La correlación lineal debe ser 0.
pca2_trans.corr()
| PC1 | PC2 | PC3 | |
|---|---|---|---|
| PC1 | 1.000000e+00 | -5.974580e-18 | -2.520275e-18 |
| PC2 | -5.974580e-18 | 1.000000e+00 | 1.426259e-15 |
| PC3 | -2.520275e-18 | 1.426259e-15 | 1.000000e+00 |
## Plot loadings
plot_loadings_heatmap(pca2,df2_pca,n_compo=3)
view_index(df2_pca, ["Guainia","Arauca"])
| DSex_p | DHom_p | DIntra_p | DTransi_p | DAsal_p | |
|---|---|---|---|---|---|
| Guainia | 85.163191 | 39.129034 | 135.800764 | 64.447820 | 202.550292 |
| Arauca | 82.745985 | 15.884274 | 316.577271 | 79.421369 | 291.827356 |
df2_pca.mean()
DSex_p 45.771800
DHom_p 7.032346
DIntra_p 139.703236
DTransi_p 81.814546
DAsal_p 223.587321
dtype: float64
fig = px.scatter(
pca2_trans,
x="PC1",
y="PC2",
text=pca2_trans.index,
hover_name=pca2_trans.index,
title="Interpretación"
)
fig.add_hline(y=0, line_color="yellow")
fig.add_vline(x=0, line_color="yellow")
biplot(pca2, df2_pca, 1, 2)
biplot(pca2,df2_pca, 1, 3)
biplot(pca2, df2_pca, 2, 3)
Ejemplo 3.#
El siguiente dataframe contiene los cambios de indices bursatiles para las 517 empresas más importantes de los EEUU. Este dataframe es muy usado en series de tiempo pues muestra el comportamiento de las empresas a través de la historia.
df_path3 = "sp500_data.csv.gz"
sp500_px = pd.read_csv(url_base+df_path3, compression='gzip', index_col=0)
sp500_px.head()
| ADS | CA | MSFT | RHT | CTSH | CSC | EMC | IBM | XRX | ALTR | ... | WAT | ALXN | AMGN | BXLT | BIIB | CELG | GILD | REGN | VRTX | HSIC | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1993-01-29 | 0.0 | 0.060124 | -0.022100 | 0.0 | 0.0 | 0.018897 | 0.007368 | 0.092165 | 0.259140 | -0.007105 | ... | 0.0 | 0.0 | 0.34716 | 0.0 | 0.04167 | 0.00000 | 0.015564 | 1.75 | 0.1250 | 0.0 |
| 1993-02-01 | 0.0 | -0.180389 | 0.027621 | 0.0 | 0.0 | 0.018889 | 0.018425 | 0.115207 | -0.100775 | 0.063893 | ... | 0.0 | 0.0 | -0.23144 | 0.0 | 0.00000 | -0.01041 | 0.007782 | 1.25 | 0.1250 | 0.0 |
| 1993-02-02 | 0.0 | -0.120257 | 0.035900 | 0.0 | 0.0 | -0.075573 | 0.029482 | -0.023041 | 0.028796 | -0.014192 | ... | 0.0 | 0.0 | -0.11572 | 0.0 | 0.00000 | 0.00000 | -0.007792 | -0.25 | 0.0000 | 0.0 |
| 1993-02-03 | 0.0 | 0.060124 | -0.024857 | 0.0 | 0.0 | -0.151128 | 0.003689 | -0.253454 | -0.043190 | -0.007105 | ... | 0.0 | 0.0 | -0.08679 | 0.0 | 0.04167 | -0.04167 | -0.038919 | -0.50 | 0.0625 | 0.0 |
| 1993-02-04 | 0.0 | -0.360770 | -0.060757 | 0.0 | 0.0 | 0.113350 | -0.022114 | 0.069862 | 0.000000 | -0.007096 | ... | 0.0 | 0.0 | 0.14465 | 0.0 | -0.04166 | -0.03126 | -0.046711 | 0.00 | 0.0625 | 0.0 |
5 rows × 517 columns
Para muestro análisis vamos a considerar las siguientes empresas:
Eitiqueta |
Nombre |
Sector |
|---|---|---|
AAPL |
Apple Inc. |
Information Technology |
MSFT |
Microsoft Corp |
Information Technology |
CSCO |
Cisco Systems |
Information Technology |
INTC |
Intel Corp |
Information Technology |
CVX |
Chevron Corp. |
Energy |
XOM |
Exxon Mobil Corp |
Energy |
SLB |
Schlumberger Ltd |
Energy |
COP |
ConocoPhillips |
Energy |
JPM |
JPMorgan Chase & Co |
Financials |
WFC |
Wells Fargo |
Financials |
USB |
U.S. Bancorp |
Financials |
AXP |
American Express Co |
Financials |
WMT |
Wal-Mart Stores |
Consumer Staples |
TGT |
Target Corp. |
Consumer Discretionary |
HD |
Home Depot |
Consumer Discretionary |
COST |
Costco Wholesale Corp. |
Consumer Staples |
Además de considerar los datos desde el 01 de enero de 2011 y hasta el final de los registros, en este caso 01 de julio del año 2015.
col_sel = sorted(["AAPL", "MSFT", "CSCO", "INTC", "CVX", "XOM", "SLB", "COP", "JPM", "WFC",
"USB", "AXP", "WMT", "TGT", "HD", "COST"])
df_pca3 = sp500_px.loc[sp500_px.index >= "2011-01-01", col_sel]
df_pca3.head()
| AAPL | AXP | COP | COST | CSCO | CVX | HD | INTC | JPM | MSFT | SLB | TGT | USB | WFC | WMT | XOM | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2011-01-03 | 0.527368 | 0.093870 | -0.336272 | -0.240605 | 0.035704 | 0.240681 | 0.099184 | -0.137211 | 0.512093 | -0.061805 | -0.325923 | 0.455646 | -0.234866 | 0.250042 | 0.294839 | 0.736805 |
| 2011-01-04 | -0.154321 | -0.431788 | -0.463161 | -0.171859 | 0.008926 | -0.584516 | -0.541005 | 0.025726 | 0.335894 | 0.132440 | -2.030049 | -0.580720 | -0.153566 | 0.000000 | 0.142951 | 0.168668 |
| 2011-01-05 | 0.597152 | 0.895406 | -0.057104 | -0.859307 | 0.169599 | 0.446985 | -0.054099 | -0.214392 | 0.689468 | 0.088294 | 1.536499 | -0.482448 | 0.198732 | 0.857284 | -0.303772 | 0.026631 |
| 2011-01-06 | -0.132850 | -0.612646 | -0.463161 | 0.249200 | 0.035706 | -0.919751 | -0.189354 | 0.085757 | 0.070713 | 0.688689 | -1.927614 | -0.786210 | -0.532962 | 0.000000 | -0.312709 | 0.248558 |
| 2011-01-07 | 0.285820 | -0.537242 | -0.006350 | -0.257788 | 0.098187 | 0.180511 | -0.036064 | -0.042878 | -0.795539 | -0.035317 | 0.931215 | -0.089345 | -0.171633 | -0.651894 | 0.169758 | 0.337329 |
Realizamos la rutina habitual.
scaler3 = StandardScaler()
scaler3.fit(df_pca3)
X_scaled3 = scaler3.transform(df_pca3)
pd.DataFrame(X_scaled3, index=df_pca3.index, columns=df_pca3.columns)
| AAPL | AXP | COP | COST | CSCO | CVX | HD | INTC | JPM | MSFT | SLB | TGT | USB | WFC | WMT | XOM | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2011-01-03 | 0.561707 | 0.110812 | -0.509688 | -0.299786 | 0.117204 | 0.252913 | 0.094034 | -0.555764 | 0.920070 | -0.213019 | -0.267647 | 0.794378 | -0.764788 | 0.630204 | 0.509947 | 0.985516 |
| 2011-01-04 | -0.107735 | -0.651798 | -0.710903 | -0.220968 | -0.008461 | -0.599033 | -0.824095 | 0.021865 | 0.596087 | 0.317332 | -1.805599 | -1.045530 | -0.513618 | -0.026284 | 0.212894 | 0.193236 |
| 2011-01-05 | 0.630238 | 1.273658 | -0.066998 | -1.009130 | 0.745557 | 0.465905 | -0.125796 | -0.829379 | 1.246217 | 0.196799 | 1.413166 | -0.871062 | 0.574786 | 2.224522 | -0.660779 | -0.004839 |
| 2011-01-06 | -0.086650 | -0.914182 | -0.710904 | 0.261778 | 0.117213 | -0.945136 | -0.319773 | 0.234680 | 0.108487 | 1.836075 | -1.713152 | -1.410346 | -1.685740 | -0.026284 | -0.678256 | 0.304644 |
| 2011-01-07 | 0.324498 | -0.804788 | 0.013486 | -0.319486 | 0.410429 | 0.190793 | -0.099932 | -0.221342 | -1.484324 | -0.140698 | 0.866905 | -0.173168 | -0.569436 | -1.737836 | 0.265322 | 0.428437 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 2015-06-25 | -1.291754 | -1.629728 | -1.482904 | -1.170435 | -0.795464 | -1.141553 | -1.482363 | -0.175690 | -1.245487 | -1.081788 | -0.596224 | 0.127481 | -0.776401 | -1.549084 | -1.220558 | -1.324935 |
| 2015-06-26 | -0.859656 | -0.444529 | 0.071124 | 0.113645 | -1.028313 | 0.376100 | -0.292015 | -1.806437 | -0.003276 | -1.109110 | 0.279190 | -0.742435 | -0.100977 | 0.236264 | 0.246246 | -0.000142 |
| 2015-06-29 | -0.869479 | -2.077787 | -0.626603 | -1.537309 | -2.145990 | -0.831825 | -1.511042 | -1.168325 | -0.898404 | -1.873597 | 0.080650 | -2.482293 | -1.583905 | -1.969155 | -0.809870 | -0.362721 |
| 2015-06-30 | -0.093671 | -0.678215 | -0.166733 | -1.537325 | -1.773440 | -1.007334 | -0.076901 | -0.813805 | -0.259013 | -1.573247 | -0.144973 | -1.825412 | -1.398532 | -0.813934 | -1.787726 | -0.795020 |
| 2015-07-01 | -0.250801 | -0.373553 | -2.323355 | 0.778636 | -0.378852 | -0.212384 | -0.593184 | -1.168318 | -0.113478 | -0.071570 | -1.886774 | 1.565507 | 0.671398 | -0.498876 | 0.480924 | -1.310987 |
1131 rows × 16 columns
## Realizamos la rutina habitual.
pca3 = PCA()
pca3.fit(X_scaled3)
PCA()In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
PCA()
## Plot de sedimentación.
sedimentacion(pca3)
Como se ve en el plot anterior, la varianza del primer componente principal es bastante grande (como suele ser el caso), pero los otros componentes principales también son significativos.
¿Cuántas compomentes principales de deben seleccionar?
## Plot loadings
plot_loadings_heatmap(pca3, df_pca3, n_compo=5)
En el heatmap anterior se muestran las cargas para las primeras 5 compomentes principales del objeto pca3.
Los loadings del primer componente principal tienen el mismo signo, esto sucede típicamente cuando las features comparten un factor común (aquí, la tendencia general del mercado de valores).
plot_loadings_bar(pca3, df_pca3, n_compo=6)
Al parecer el segundo componente captura los cambios de precio en aquellas acciones del sector de energía en comparación con las otras acciones.
El tercer componente es principalmente un contraste en los movimientos de Apple y Cost.
El cuarto componente contrasta los movimientos de Schlumberger (SLB) al resto de acciones energéticas.
El quinto componente está mayoritariamente dominado por empresas financieras.
biplot(pca3, df_pca3, 1, 5)
biplot1(pca3,df_pca3)
Conclusiones.#
PCA es una técnica lineal que busca reducir la dimensionalidad del dataset buscando optimizar la perdida de información presente en la variabilidad.
Una desventaja de PCA es que los dos ejes del primer plano factorial a menudo no son muy fáciles de interpretar. Esto debido a que las componentes principales corresponden a direcciones en los datos originales, dado que son combinaciones de las features originales.
Existen técnicas más modernas para realizar la reducción de dimensionalidad en el dataset, algunas las desarrollaremos más adelante en el curso.
Tarea 2#
Realizar un análisis de compomentes principales de la tabla de los resultados de las pruebas ICFES 2019-2.
